library(tidyverse)凡例の設定のところでご紹介した新しい関数はscale_color_discreteと、scale_fill_discreteの二つですが、中身の設定がすこしごちゃごちゃしていたと思いますので、ここで演習問題を解きながら身に着けていきましょう。
利用するのはmsleepデータです、まず、
base_graph <- ggplot(msleep) +
geom_point(aes(x=log(bodywt), y=log(brainwt), color=vore))
base_graphWarning: Removed 27 rows containing missing values (`geom_point()`).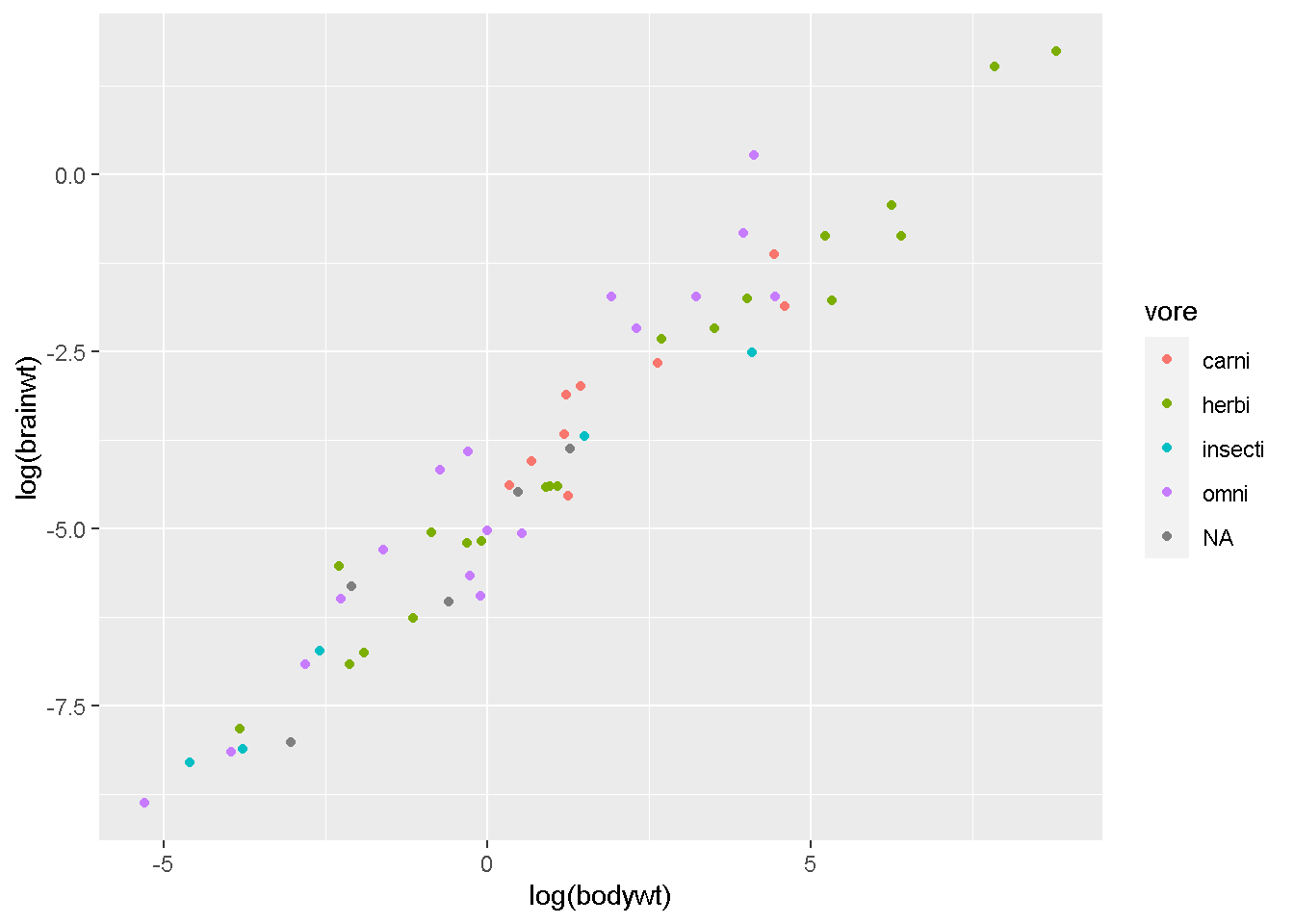
このbase_graphがあるとして、これのラベルと凡例を「キレイ」にしてみてください。(尚、x軸、y軸ともに、log関数を理容して対数にしてあります。対数にしないと)
ggplot(msleep)+geom_point(aes(x=bodywt,y=brainwt))Warning: Removed 27 rows containing missing values (`geom_point()`).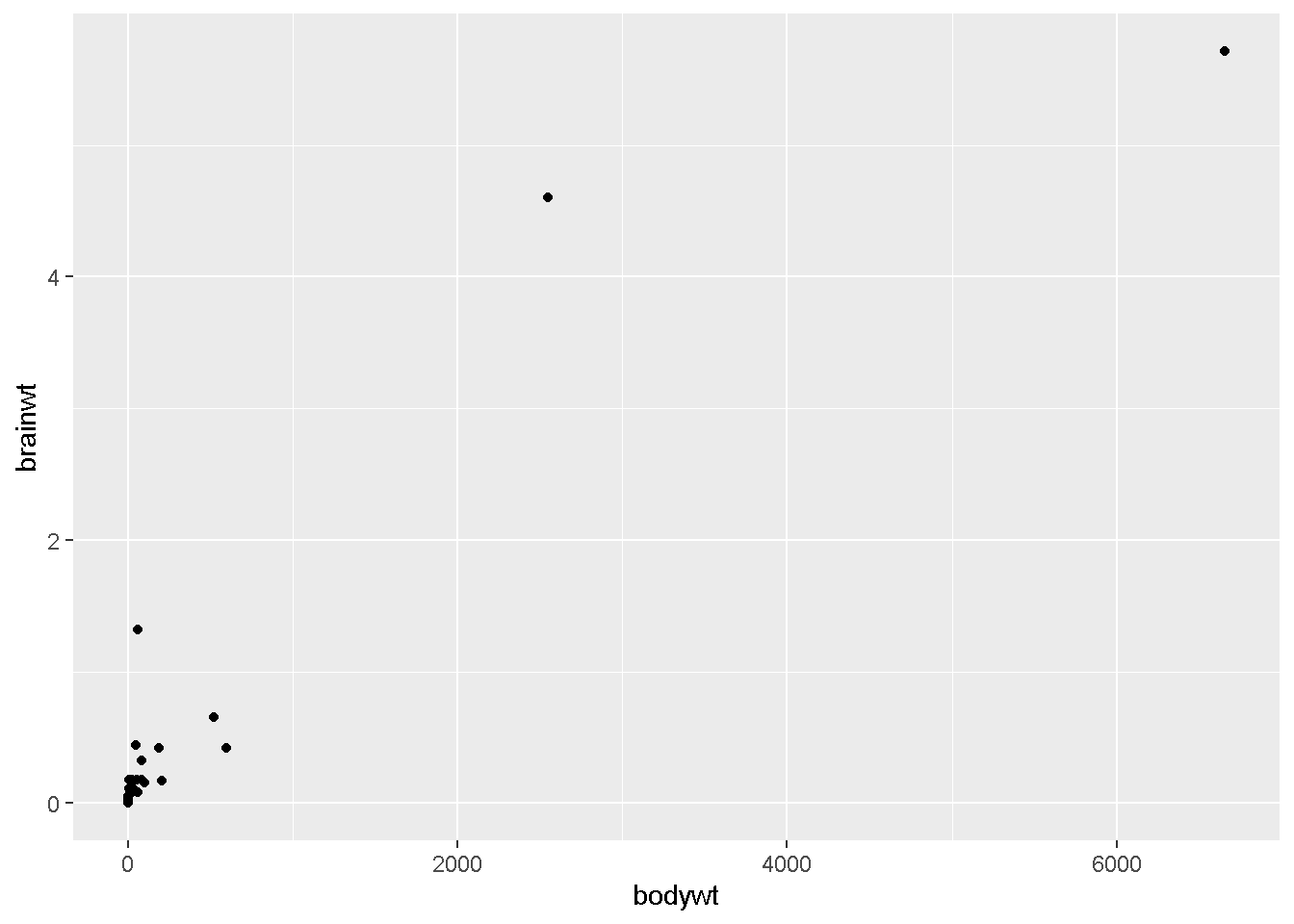
こんな感じで左下に「ぐしゃっ」となるためです。
では、
base_graphWarning: Removed 27 rows containing missing values (`geom_point()`).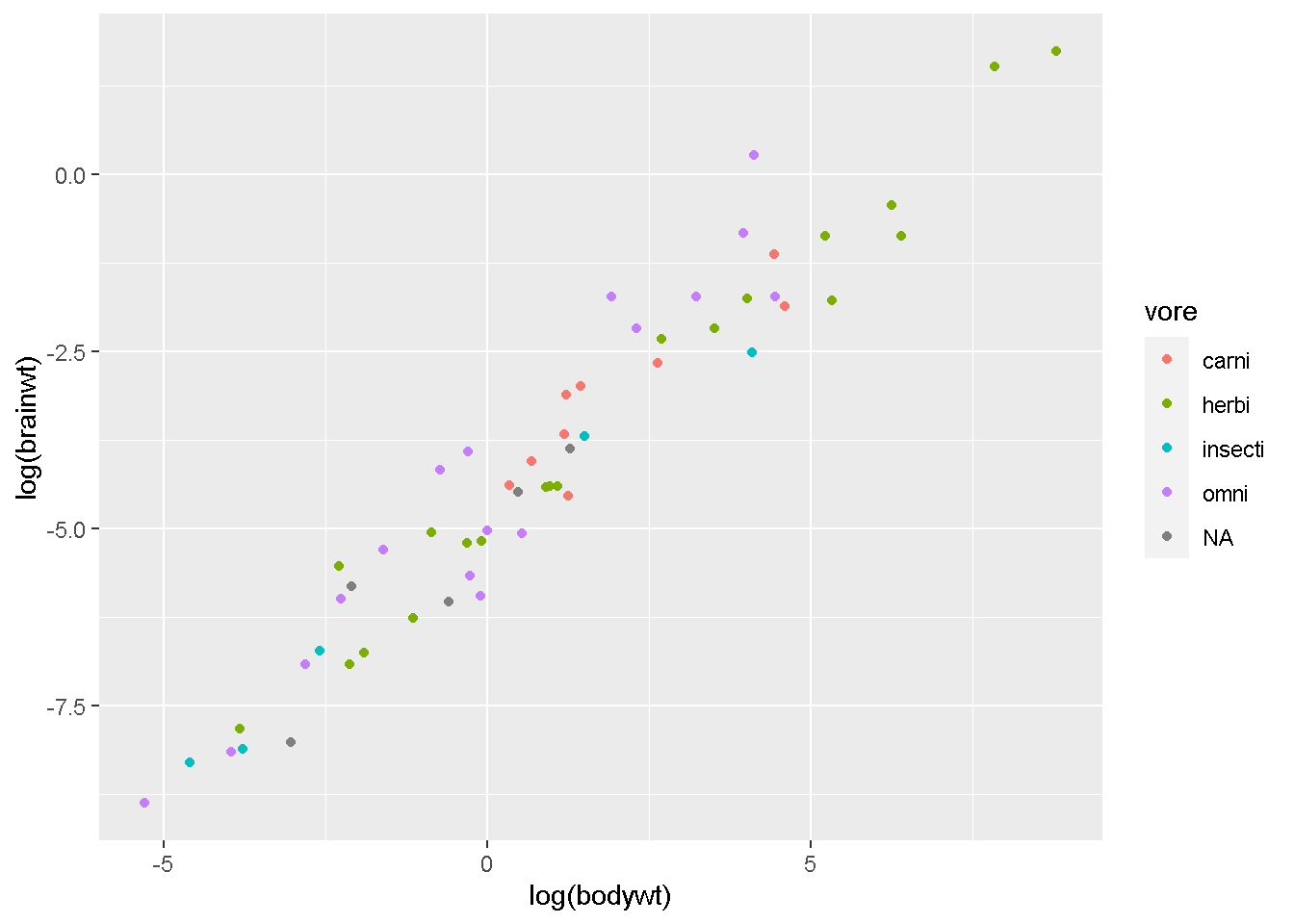
のタイトルを 体重と脳の重さの関係
- x軸のラベルを 体重[log(kg)]
- y軸のラベルを 脳の重さ[log(kg)]
- 凡例のタイトルを 食性
- 凡例のラベルをそれぞれ
- carni 肉食
- herbi 草食
- insecti 虫食
- onmi 雑食
- NA その他
として、carni insecti hrebi omni NAの順番に並び替えてみてください。 それでは、動画を止めてどうぞ。 できましたか?
タイトルとラベルをまず設定するのはlabs関数、でしたね?
base_graph +
labs(title = "体重と脳の重さの関係",
x = "体重[log(kg)]", y = "脳の重さ[log(kg)]")Warning: Removed 27 rows containing missing values (`geom_point()`).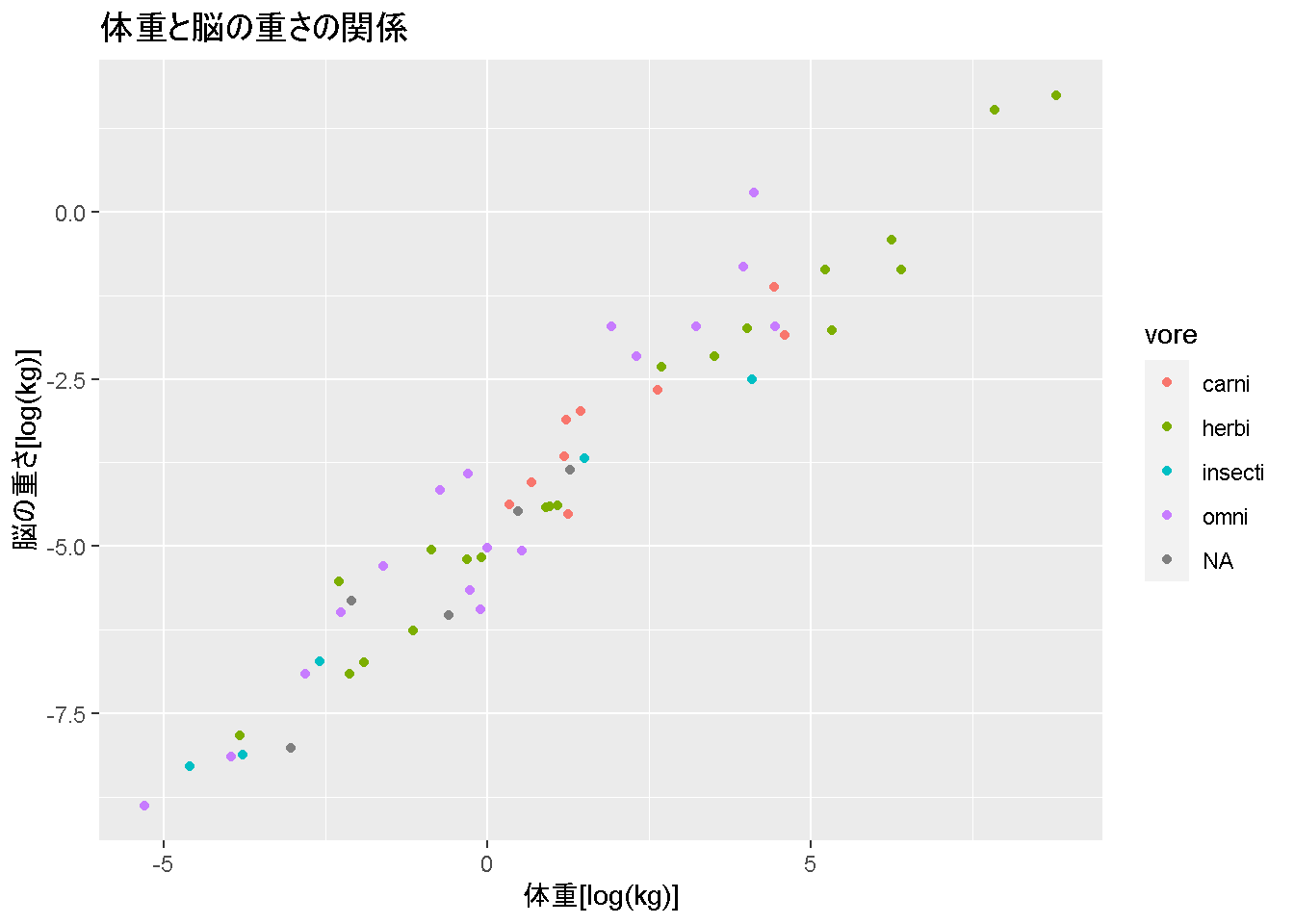
それで、凡例を設定するためには、ここで、scale_color_discreteか、scale_fill_discreteのどちらかに設定を行ってあげる必要がありますがどちらを利用するか、判断できましたでしょうか?
今回の場合は、base_graphは、
ggplot(msleep) +
geom_point(aes(x=log(bodywt), y=log(brainwt), color=vore))color=voreと、colorを利用して凡例を描画 しているため、次のようなスクリプトが正解になります
base_graph +
scale_color_discrete(
name="食性"
)Warning: Removed 27 rows containing missing values (`geom_point()`).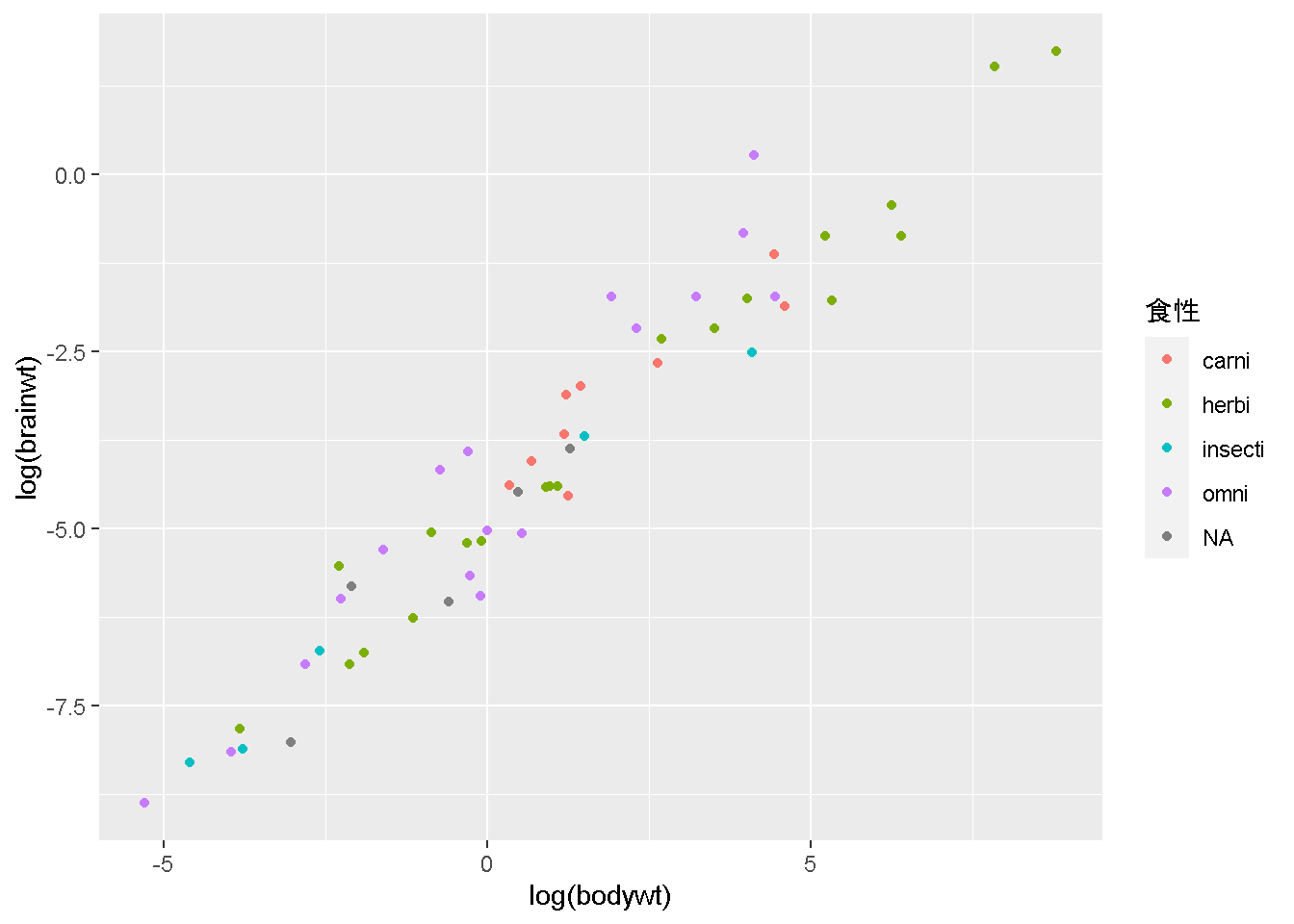
それで、あとはラベルと並び順です。一応、NAが文字列のNAなのか欠損のNAなのかは確認したいので、
str(msleep$vore) chr [1:83] "carni" "omni" "herbi" "omni" "herbi" "herbi" "carni" NA ...summary(msleep$vore) Length Class Mode
83 character character levels(as.factor(msleep$vore))[1] "carni" "herbi" "insecti" "omni" もともとのデータが文字列型で、本当に欠損しているみたいです。ggplotでは、文字列型は因子型と同様の扱いになってカテゴリー変数とみなされるため、
narabi <- c("carni","insecti","herbi","omni", NA)
hyouji <- c("肉食","虫食","草食","雑食","その他")ここでnarabiのNAは”NA”でなくて、NAであることに注意しておいてください。
NA[1] NA"NA"[1] "NA"は明確に違います。NAは欠損を表すもので
str(NA) #ロジカル型 logi NAstr("NA") #文字列型 chr "NA"です。
それで、作成した変数を利用して、
base_graph +
scale_color_discrete(
name = "食性",
breaks = narabi,
labels = hyouji
)Warning: Removed 27 rows containing missing values (`geom_point()`).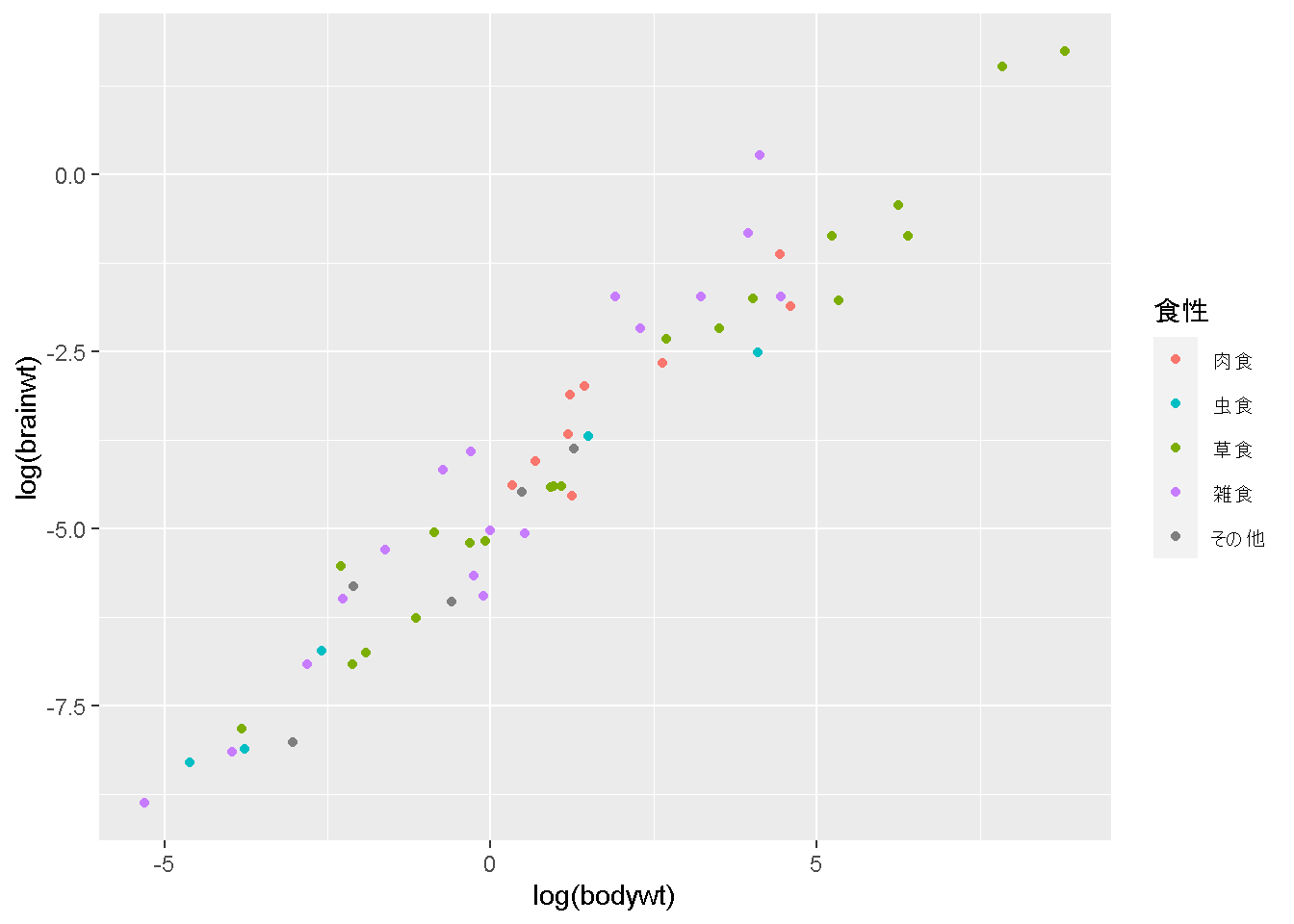
となります。
いかがでしょうか?
あともう1問やっておきましょう。
dat <- tibble(a=c(1,2,3),b=c(1,2,3),n=c("a","b","b"))
ggplot(dat)+geom_col(aes(x=a,y=b,fill=n))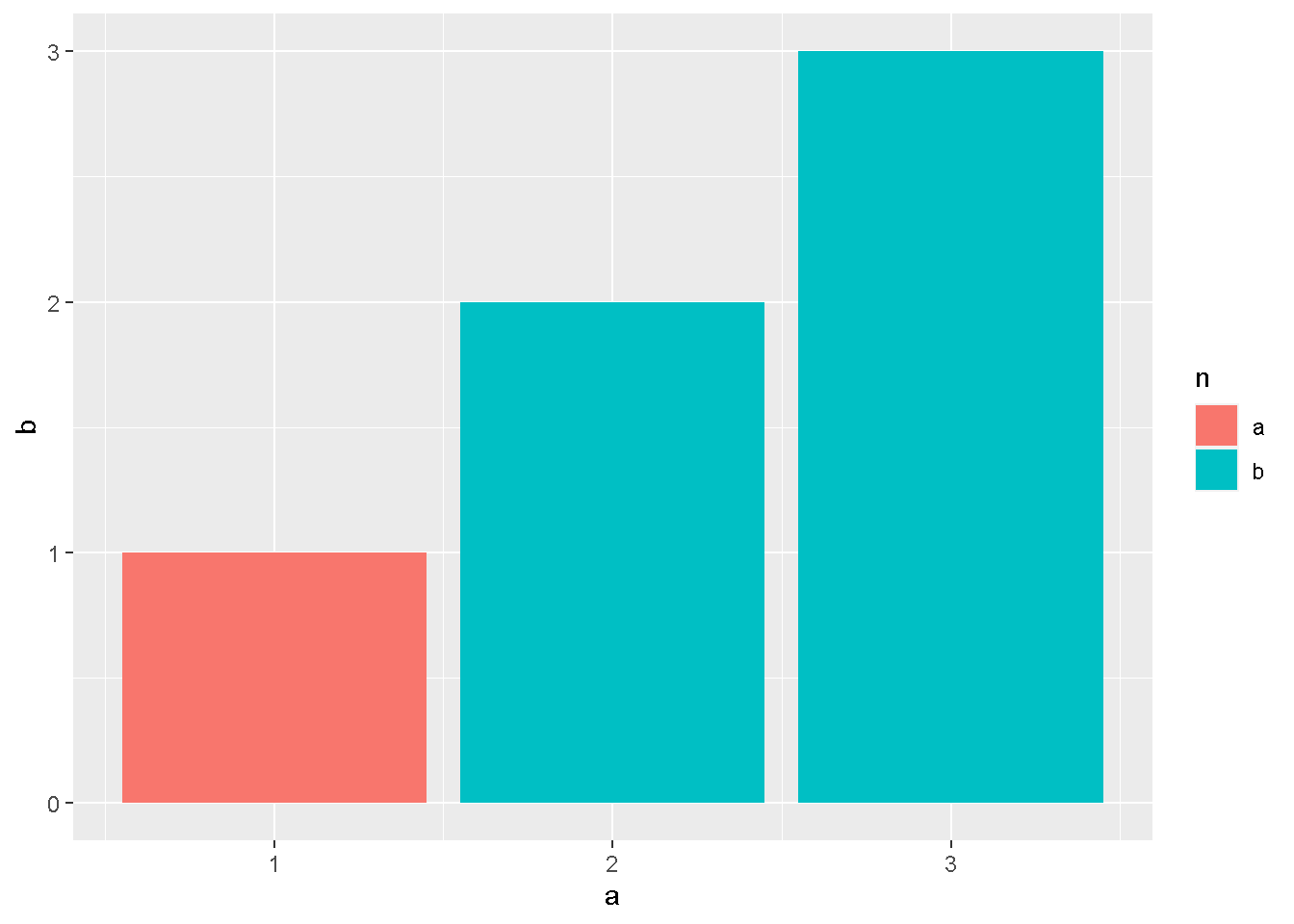
geom_colは初めてでてくるgeomですが、棒グラフを集計なしで作成するgeomです。geom_barだと、x軸の指定をすると勝手に集計してくれていましたが、geom_colはy軸の値の指定も必要です。
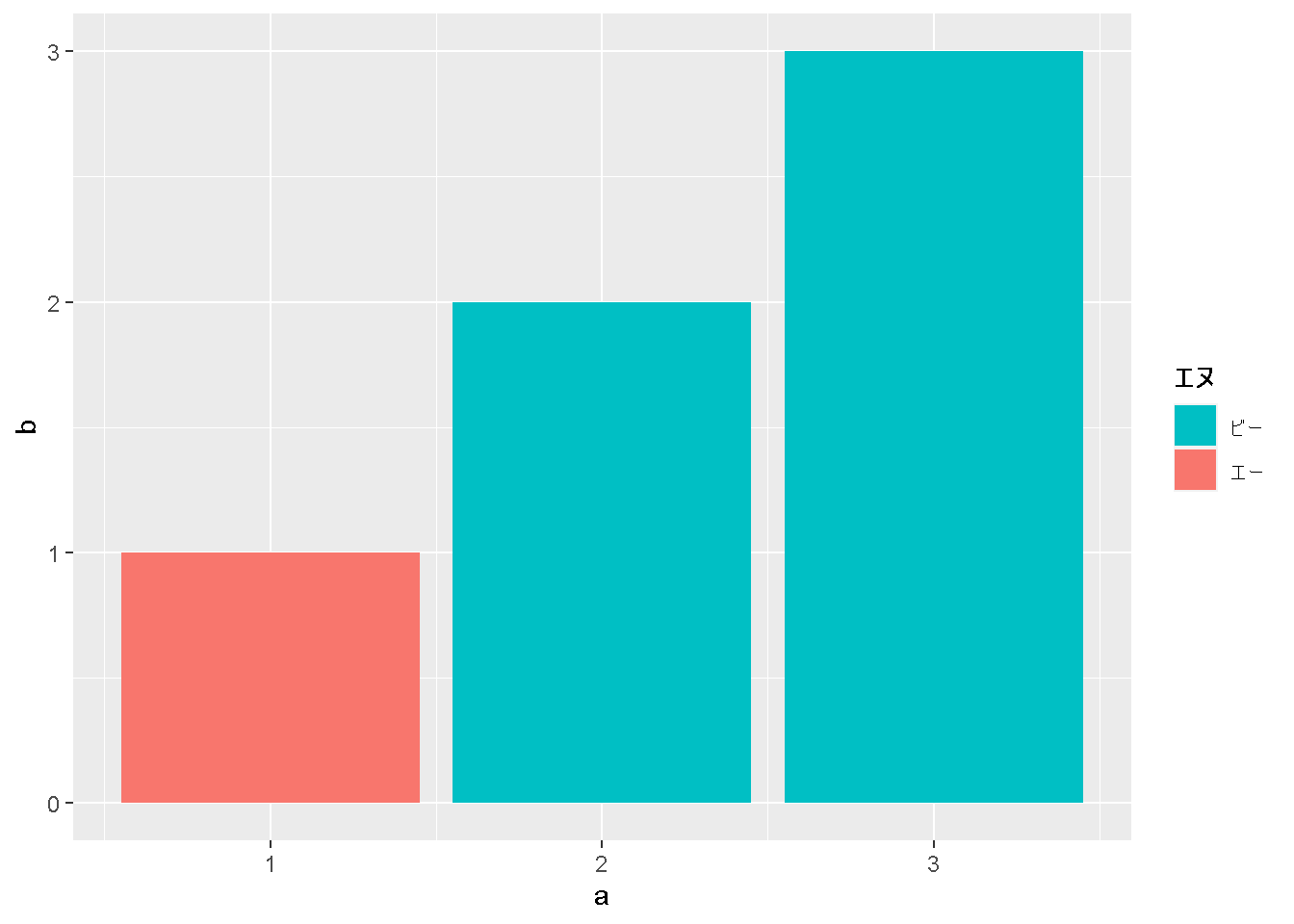
こんなグラフ、凡例のタイトルをエヌ、凡例の並びをa->bではなく、b->aにして、aはエー、bはビーというラベルを付けてみてください。
できましたか?
ggplot(dat)+
geom_col(aes(x=a,y=b,fill=n)) +
scale_fill_discrete(
name="エヌ",
breaks=c("b","a"),
labels=c("ビー","エー")
)
こんな感じで自分でデータを作って関数の動きを確認できるようになると、学習速度が上がることと、将来、関数を作成した時に意図した通りに動くかの確認をしたりするときに非常に役立つのですきをみて取り組んでみてください。
それでは、次はggplotの基礎の最後、テーマの設定について解説していきます。