library(tidyverse)
library(readxl)
dat_a <- read_excel("data/food_poisoning2020.xls",
range = "A3:W36",
col_names = FALSE,
sheet="④病因物質別発生状況")
dat_b <- read_excel("data/food_poisoning2020.xls",
range = "A39:T72",
col_names = FALSE,
sheet="④病因物質別発生状況")
row1 <- dat_a %>% slice(1) %>% as_vector()
row2 <- dat_a %>% slice(2) %>% as_vector()
col_a <- tibble(r1 = row1, r2 = row2) %>%
fill(r1, r2) %>%
replace_na(list(r2="")) %>%
unite("col_a",r1, r2, sep="_")
col_a$col_a[1] <- "cause_1"
col_a$col_a[2] <- "cause_2"
dat_a2 <- dat_a %>%
setNames(col_a$col_a) %>%
slice(-c(1:2)) %>%
fill(cause_1) %>%
filter(!is.na(cause_2)) %>%
pivot_longer(
cols = !c(cause_1, cause_2),
names_to = c("month","type"),
values_to = "val",
names_sep = "_"
) %>%
filter(month != "総数")
row1 <- dat_b %>% slice(1) %>% as_vector()
row2 <- dat_b %>% slice(2) %>% as_vector()
col_b <- tibble(r1 = row1, r2 = row2) %>%
fill(r1, r2) %>%
replace_na(list(r2="")) %>%
unite("col_b",r1, r2, sep="_")
col_b$col_b[1] <- "cause_1"
col_b$col_b[2] <- "cause_2"
dat_b2 <- dat_b %>%
setNames(col_b$col_b) %>%
slice(-c(1:2)) %>%
fill(cause_1) %>%
filter(!is.na(cause_2)) %>%
pivot_longer(
cols = !c(cause_1, cause_2),
names_to = c("month","type"),
values_to = "val",
names_sep = "_"
) %>%
filter(month != "総数")ひとつ前の動画で作成した二つのデータ、結合しましょう。
ここでの結合は二つのデータの列同士を結合するやり方になり、join系の関数を利用しません。
join系の関数は、
A <- A B <- B C <- C
こうですが、
ここで行いたい結合は、
A B C A B C
という縦方向の結合です。この結合を行うのは、
test1 <- tibble(A = c(1:3), B = c(11:13))
test2 <- tibble(A = c(4:6), B = c(14:16))
test1# A tibble: 3 × 2
A B
<int> <int>
1 1 11
2 2 12
3 3 13test2# A tibble: 3 × 2
A B
<int> <int>
1 4 14
2 5 15
3 6 16bind_rows(test1, test2)# A tibble: 6 × 2
A B
<int> <int>
1 1 11
2 2 12
3 3 13
4 4 14
5 5 15
6 6 16この、bind_rows関数です。
ということで、
dat_fin <- bind_rows(dat_a2, dat_b2)
View(dat_fin)くっつきました。
後は、cause_1とcause_2の余分な空白、
dat_fin$cause_1 %>% unique()[1] "細 菌"
[2] "ウ イ ル ス"
[3] "寄 生 虫"
[4] "自 然 毒"dat_fin$cause_2 %>% unique() [1] "サ ル モ ネ ラ 属 菌" "ぶ ど う 球 菌"
[3] "ボ ツ リ ヌ ス 菌" "腸 炎 ビ ブ リ オ"
[5] "腸管出血性大腸菌(VT産生)" "その他の病原大腸菌"
[7] "ウ エ ル シ ュ 菌" "セ レ ウ ス 菌"
[9] "エルシニア・エンテロコリチカ" "カンピロバクター・ジェジュニ/コリ"
[11] "ナ グ ビ ブ リ オ" "コ レ ラ 菌"
[13] "赤 痢 菌" "チ フ ス 菌"
[15] "パ ラ チ フ ス A 菌" "そ の 他 の 細 菌"
[17] "ノ ロ ウ イ ル ス" "その他のウイルス"
[19] "ク ド ア" "サルコシスティス"
[21] "ア ニ サ キ ス" "その他の寄生虫"
[23] "植 物 性 自 然 毒" "動 物 性 自 然 毒" を削除しておきましょう。
str_view_all( unique(dat_fin$cause_1), " ")Warning: `str_view()` was deprecated in stringr 1.5.0.
ℹ Please use `str_view_all()` instead.[1] │ 細<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>菌
[2] │ ウ<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>イ<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>ル<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>ス
[3] │ 寄<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}> 生 <{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>虫
[4] │ 自<{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}> 然 <{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}><{\u3000}>毒str_view_all( unique(dat_fin$cause_2), " ") [1] │ サ<{\u3000}>ル<{\u3000}>モ<{\u3000}>ネ<{\u3000}>ラ<{\u3000}>属<{\u3000}>菌
[2] │ ぶ<{\u3000}><{\u3000}>ど<{\u3000}><{\u3000}>う<{\u3000}><{\u3000}>球<{\u3000}><{\u3000}>菌
[3] │ ボ<{\u3000}>ツ<{\u3000}>リ<{\u3000}>ヌ<{\u3000}>ス<{\u3000}>菌
[4] │ 腸<{\u3000}>炎<{\u3000}>ビ<{\u3000}>ブ<{\u3000}>リ<{\u3000}>オ
[5] │ 腸管出血性大腸菌(VT産生)
[6] │ その他の病原大腸菌
[7] │ ウ<{\u3000}>エ<{\u3000}>ル<{\u3000}>シ<{\u3000}>ュ<{\u3000}>菌
[8] │ セ<{\u3000}><{\u3000}>レ<{\u3000}><{\u3000}>ウ<{\u3000}><{\u3000}>ス<{\u3000}><{\u3000}>菌
[9] │ エルシニア・エンテロコリチカ
[10] │ カンピロバクター・ジェジュニ/コリ
[11] │ ナ<{\u3000}>グ<{\u3000}>ビ<{\u3000}>ブ<{\u3000}>リ<{\u3000}>オ
[12] │ コ<{\u3000}><{\u3000}>レ<{\u3000}><{\u3000}>ラ<{\u3000}><{\u3000}>菌
[13] │ 赤<{\u3000}><{\u3000}><{\u3000}><{\u3000}>痢<{\u3000}><{\u3000}><{\u3000}><{\u3000}>菌
[14] │ チ<{\u3000}><{\u3000}>フ<{\u3000}><{\u3000}>ス<{\u3000}><{\u3000}>菌
[15] │ パ ラ チ フ ス A 菌
[16] │ そ の 他 の 細 菌
[17] │ ノ<{\u3000}>ロ<{\u3000}>ウ<{\u3000}>イ<{\u3000}>ル<{\u3000}>ス
[18] │ その他のウイルス
[19] │ ク<{\u3000}><{\u3000}><{\u3000}>ド<{\u3000}><{\u3000}><{\u3000}>ア
[20] │ サルコシスティス
... and 4 moredat_fin <- dat_fin %>%
mutate(
cause_1 = str_remove_all(cause_1," "),
cause_2 = str_remove_all(cause_2," ")
)また、valは、「-」が欠損値として利用されているので置き換えておきましょう。
dat_fin <- dat_fin %>%
mutate(val = na_if(val,"-")) %>%
mutate(val = as.numeric(val))
View(dat_fin)以上!キレイな形になりました。
グラフ化してみましょう。
gdat <- dat_fin %>%
filter(cause_2 == "ノロウイルス")
ggplot(gdat) +
geom_col(aes(x = month, y = val)) +
facet_wrap(~type)Warning: Removed 18 rows containing missing values (`position_stack()`).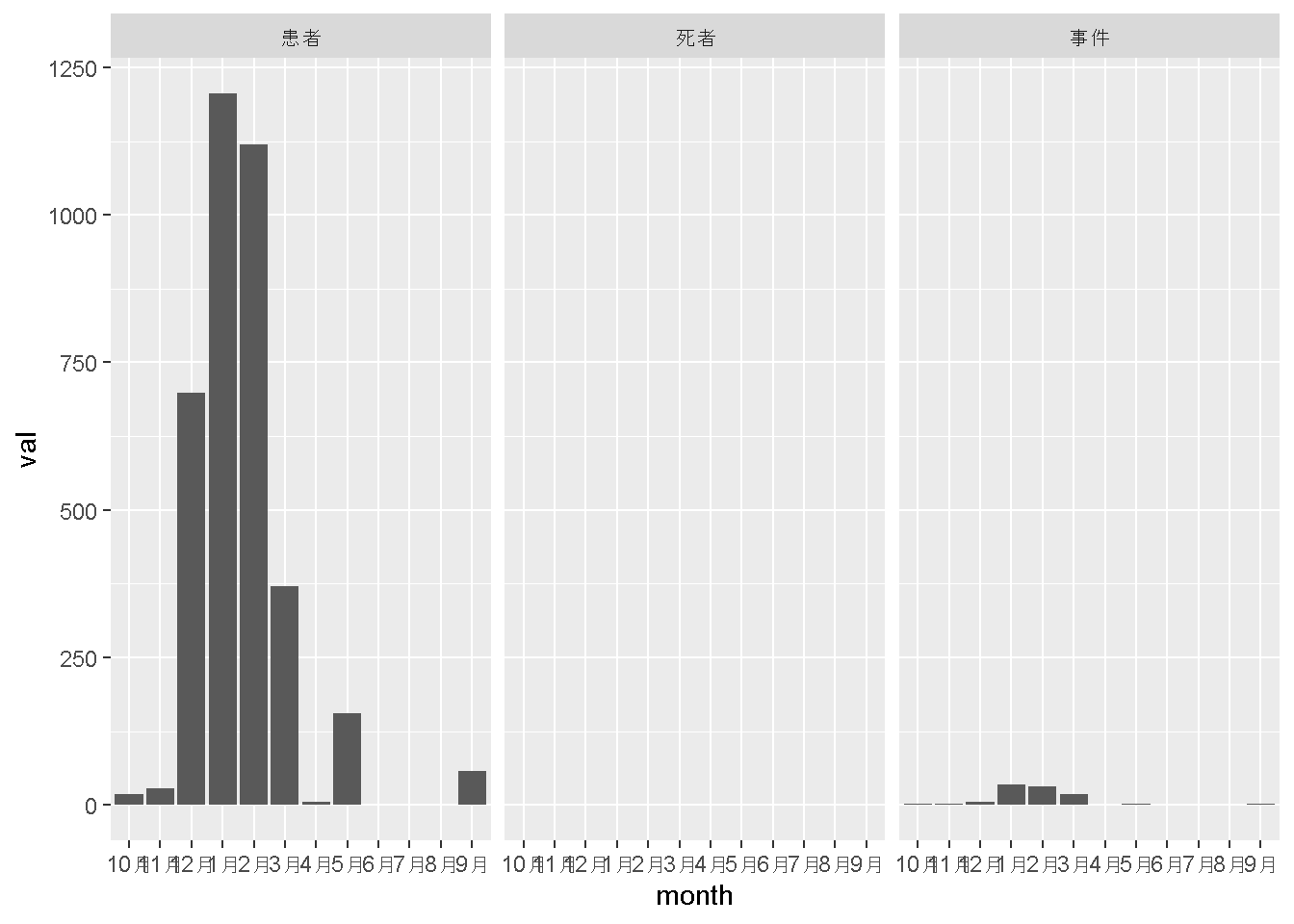
monthの並びが10月、11月、12月、1月と並んでいるのでここの並びを整えましょう。
因子型にすると並びを調整することが可能です。
month_label <- c("1月","2月","3月","4月","5月","6月",
"7月","8月","9月","10月","11月","12月")
dat_fin <- dat_fin %>%
mutate(month = factor(month,
levels=month_label,
labels=month_label))
gdat <- dat_fin %>%
filter(cause_2 == "ノロウイルス")
ggplot(gdat) +
geom_col(aes(x = month, y = val)) +
facet_wrap(~type)Warning: Removed 18 rows containing missing values (`position_stack()`).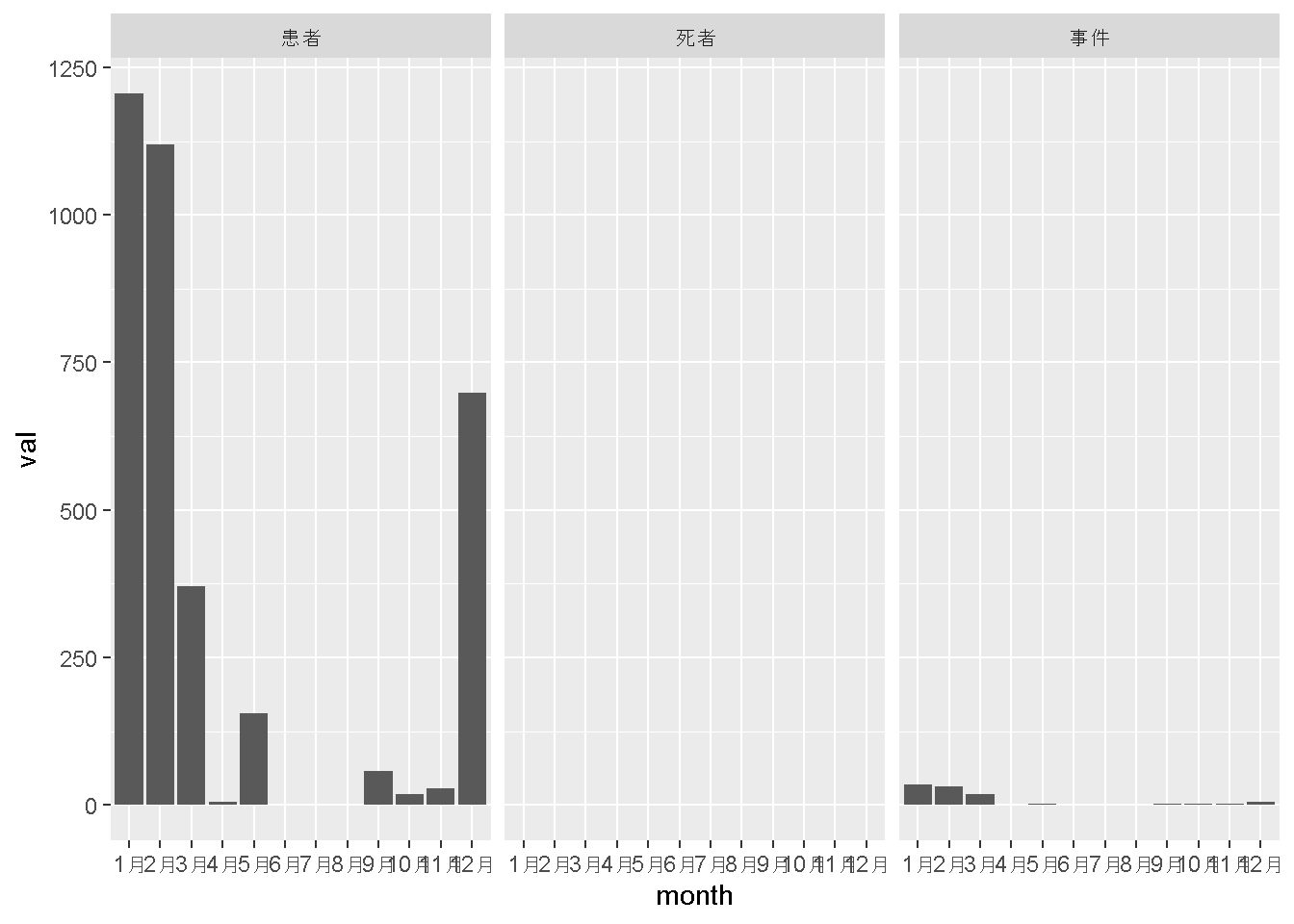
うまくならびましたね!
以上、実際のデータをインポート、加工してグラフ化する一連の流れをみてみました。
解説しながらなので、スクリプト量が多く感じるかもしれませんが、後から解説する「関数化」を利用すれば、同じ形のデータを1行で処理できたりするので、引き続きお付き合いください。
おつかれさまでした。
ここまでの加工ができるようになれば、思いどおりにデータの形を変形することができるようになっているはずです。データのインポート、可視化、データ加工の3つの手順をストレスなくできるようになれば、あなたが行いたいデータ分析はほぼ8割が終了しているという格言もあったりします。
残りのセクションで、 * データの集計を行う方法 * 分析結果を共有するときに有用なレポート作成
の知識をお伝えいたします。