library(tidyverse)
library(lubridate)
plot_id <- function(.data,tgt_id){
gdat <- .data %>%
filter(id %in% tgt_id) %>%
mutate(row_n = n():1) %>%
mutate(points = map2(start,end, ~{.x:.y})) %>%
select(id,med,row_n,points) %>%
unnest(c(points)) %>%
mutate(points = as_date(points))
ggplot(gdat) +
geom_point(aes(x = points, y = as.factor(row_n), color = med)) +
scale_y_discrete(labels=NULL) +
facet_wrap(~id, scales = "free")
}
compare_plot <- function(.data1, .data2, tgt_id){
pre_graph <- plot_id(.data1, tgt_id)
post_graph <- plot_id(.data2, tgt_id)
cowplot::plot_grid(pre_graph, post_graph, nrow=2)
}
dat <- read_csv("data/time.csv")
dat_fin <- dat %>%
arrange(id,med,start) %>% #ここにmed,startを追加
mutate(interv = interval(start, end)) %>% #同じ
group_by(id, med) %>% #idとmedでグループ化
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_edgealign = int_aligns(interv, lag(interv)),
) %>%
mutate(prev_oa = prev_overlap | prev_edgealign) %>%
replace_na(list(prev_oa = FALSE)) %>%
mutate(presc_id = cumsum(!prev_oa)) %>%
group_by(id, med, presc_id) %>% #グループを作り直し。id,medを追加
summarise(start = min(start), end = max(end)) %>%
ungroup() %>%
arrange(id,med,start) #並び替えておきます。それでは、93番のデータが想定とちがった理由を確認しておきます
これは、
compare_plot(dat,dat_fin,93)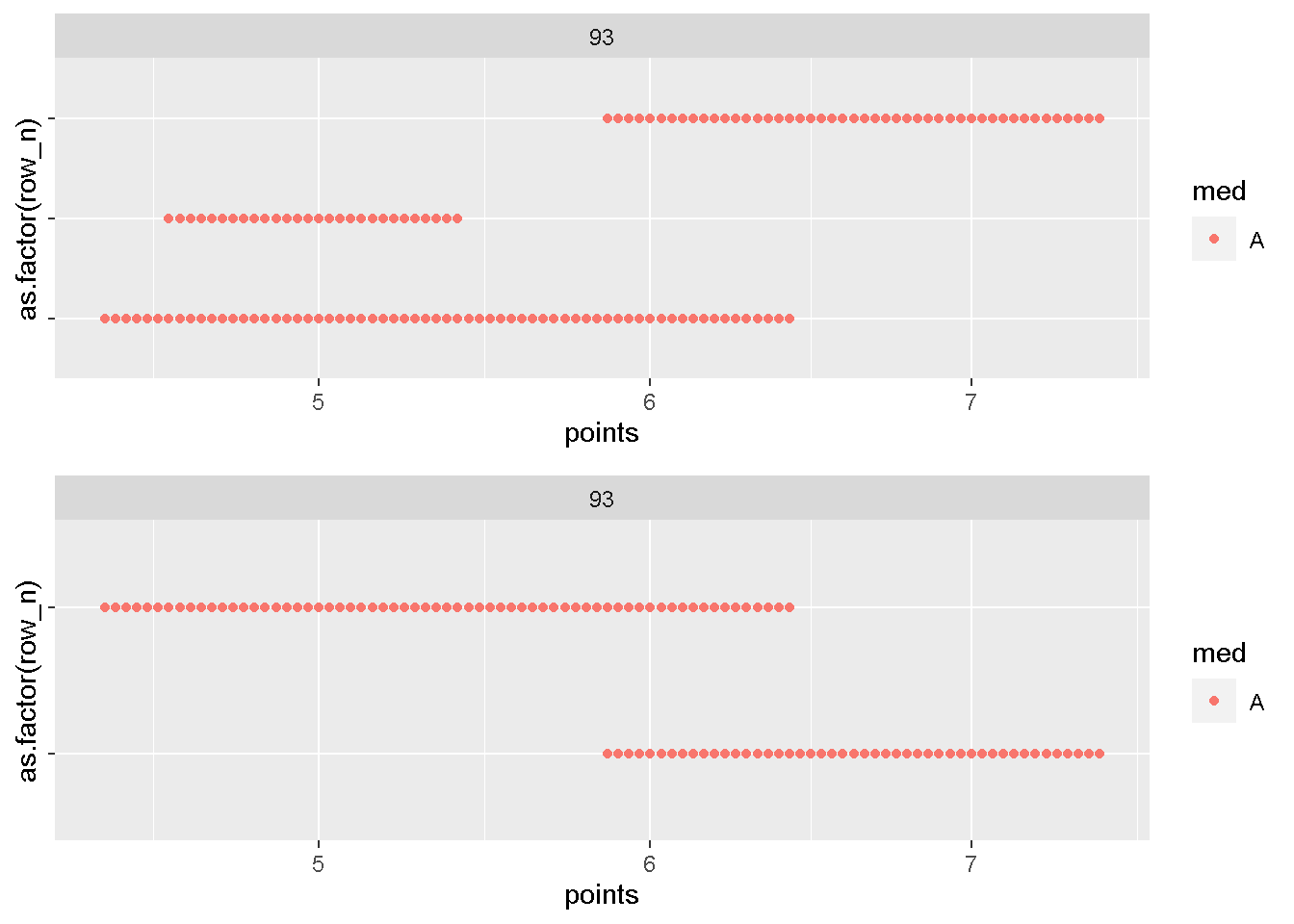
prev_overlap cumsum(!prev_oa)|——————-| FALSE 1 |—–| TRUE 1 |————–| FALSE 2
このようなパターンとなっているため、処理をした結果が、
|——————-|
|————–|
こうなっていて、処理自体は正しく終わっているのですが、うまくくっついていないケースです。
これへの対応は色々と考えられますが、ここでは、処理を複数回繰り返して対応しましょう。上の形に対して同じ処理を繰り返すと、最終的には
|—————————-|
こうなるはずです。
「処理」を行数の変化がなくなるまで複数回実行してあげて全部の行を一つにまとめましょう。
できますか?
dat_n1 <- dat %>%
arrange(id,med,start) %>%
mutate(interv = interval(start, end)) %>%
group_by(id, med) %>%
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_nextday = int_overlaps(interv, int_shift(lag(interv),days(1))),
) %>%
mutate(prev_oa = prev_overlap | prev_nextday) %>%
replace_na(list(prev_oa = FALSE)) %>%
mutate(presc_id = cumsum(!prev_oa)) %>%
group_by(id, med, presc_id) %>%
summarise(start = min(start), end = max(end)) %>%
ungroup() %>%
select(id, med, start, end) `summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.これが1回目の処理です。
行数の変化は、
nrow(dat)[1] 400nrow(dat_n1)[1] 342ですね。
もう一度。
dat_n2 <- dat_n1 %>%
arrange(id,med,start) %>%
mutate(interv = interval(start, end)) %>%
group_by(id, med) %>%
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_nextday = int_overlaps(interv, int_shift(lag(interv),days(1))),
) %>%
mutate(prev_oa = prev_overlap | prev_nextday) %>%
replace_na(list(prev_oa = FALSE)) %>%
mutate(presc_id = cumsum(!prev_oa)) %>%
group_by(id, med, presc_id) %>%
summarise(start = min(start), end = max(end)) %>%
ungroup() %>%
select(id, med, start, end) `summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.すると、
nrow(dat)[1] 400nrow(dat_n1)[1] 342nrow(dat_n2)[1] 3411行減りました。ということはこれ以上変化しないはずですが、
念のため、
dat_n3 <- dat_n2 %>%
arrange(id,med,start) %>%
mutate(interv = interval(start, end)) %>%
group_by(id, med) %>%
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_nextday = int_overlaps(interv, int_shift(lag(interv),days(1))),
) %>%
mutate(prev_oa = prev_overlap | prev_nextday) %>%
replace_na(list(prev_oa = FALSE)) %>%
mutate(presc_id = cumsum(!prev_oa)) %>%
group_by(id, med, presc_id) %>%
summarise(start = min(start), end = max(end)) %>%
ungroup() %>%
select(id, med, start, end) `summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.nrow(dat)[1] 400nrow(dat_n1)[1] 342nrow(dat_n2)[1] 341nrow(dat_n3)[1] 341はい。確かに変化していないので問題なく結合できているはずです。
93番を見てあげると、
compare_plot(dat,dat_n1,93)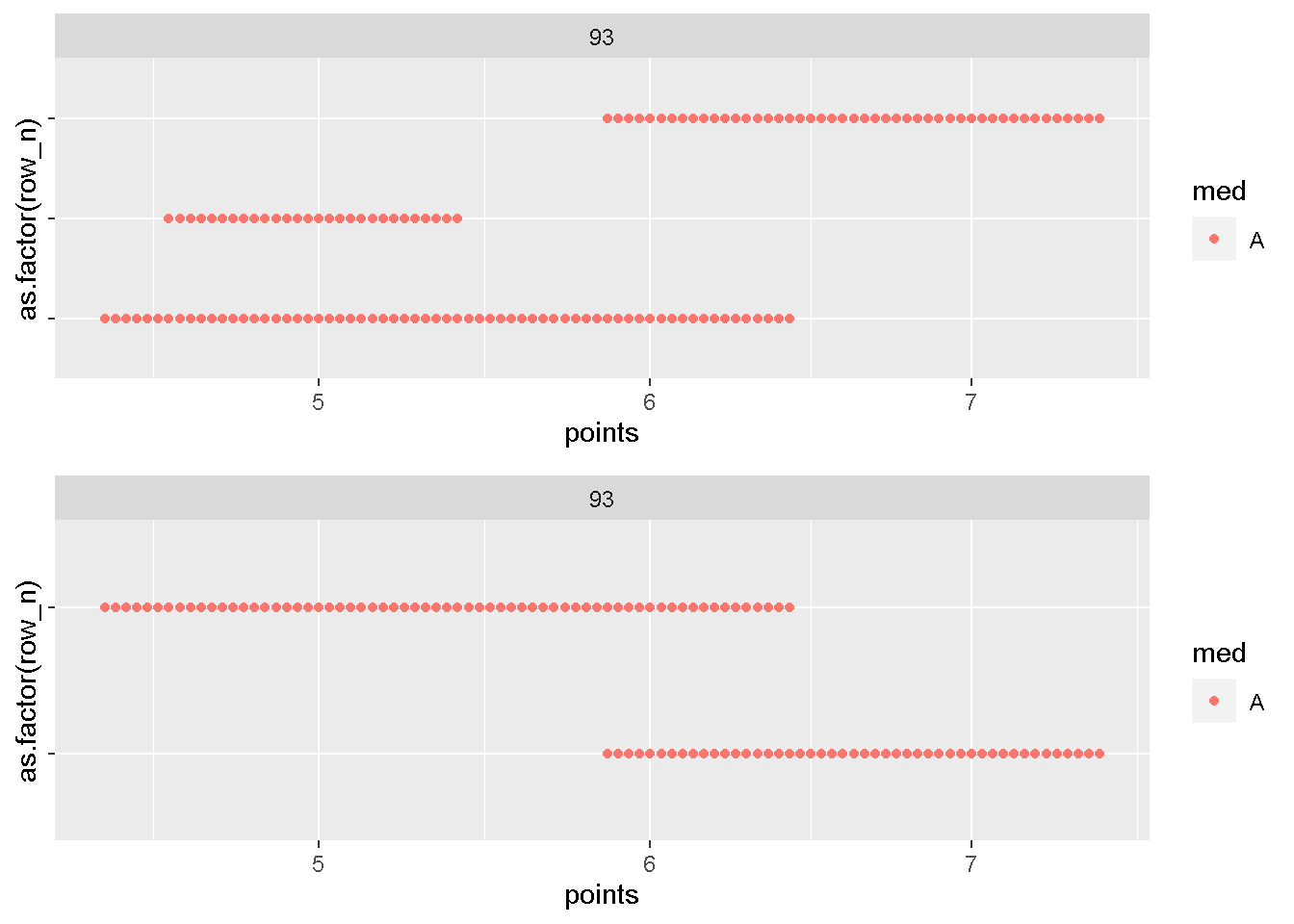
compare_plot(dat_n1,dat_n2,93)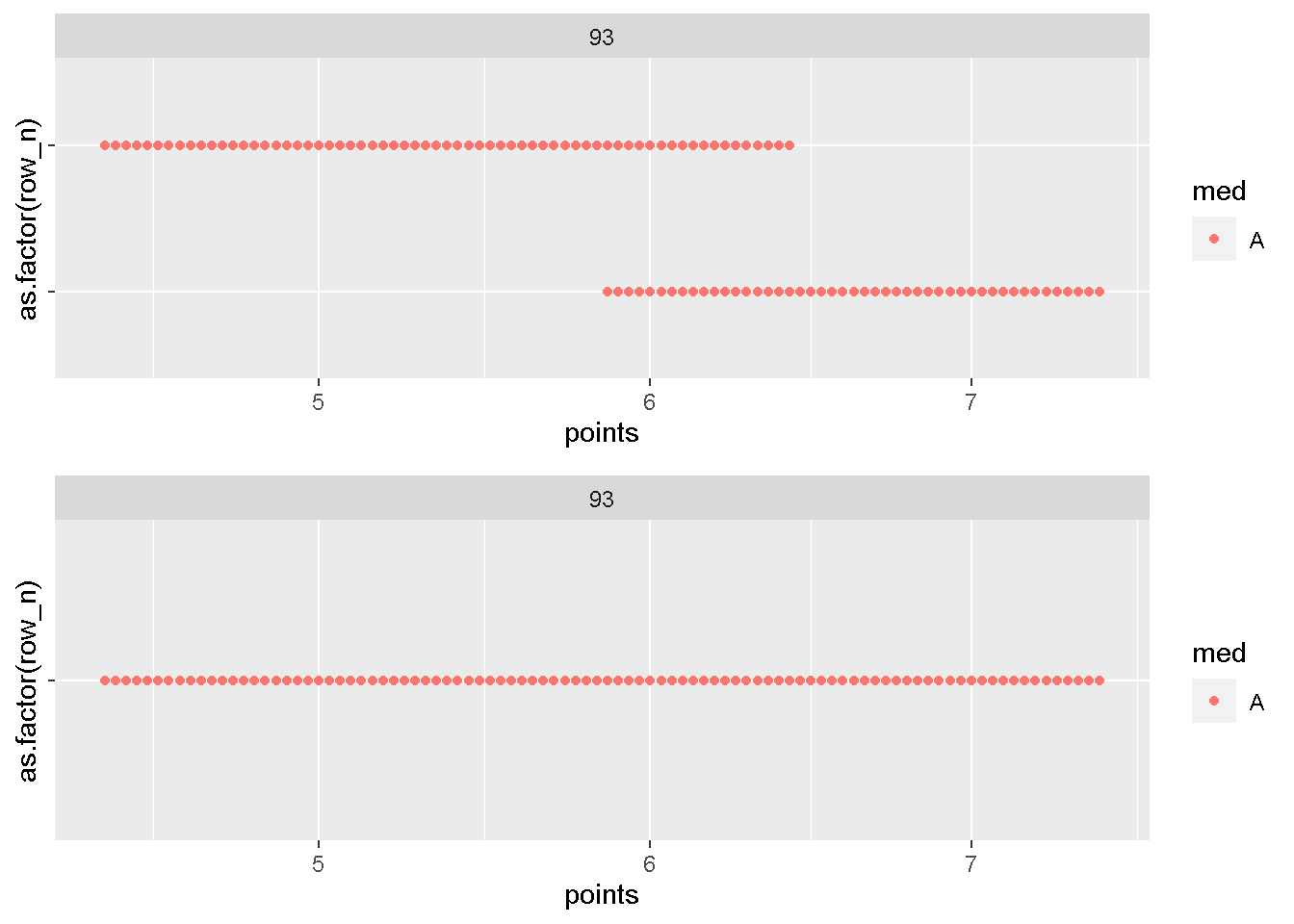
確かに結合されていますね?
この処理方法、時間もかかるし面倒なので、本来であれば
- 繰り返し処理
- 関数化
などで対応しますが、少し抽象度が高い概念となるため、本コースでは以下に例示をするだけにとどめます。
もっと勉強してみたいという方は、別のコースを確認ください。
例示:
関数を作成する
process_data <- function(.data){
fin <- .data %>%
arrange(id,med,start) %>%
mutate(interv = interval(start, end)) %>%
group_by(id, med) %>%
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_nextday = int_overlaps(interv, int_shift(lag(interv),days(1))),
) %>%
mutate(prev_oa = prev_overlap | prev_nextday) %>%
replace_na(list(prev_oa = FALSE)) %>%
mutate(presc_id = cumsum(!prev_oa)) %>%
group_by(id, med, presc_id) %>%
summarise(start = min(start), end = max(end)) %>%
ungroup() %>%
select(id, med, start, end)
return(fin)
}繰り返し処理を行う
iter_data <- dat
prerows <- nrow(iter_data)
postrows <- 0
while(postrows != prerows){
prerows <- nrow(iter_data)
print(prerows)
iter_data <- process_data(iter_data)
postrows <- nrow(iter_data)
print(postrows)
}[1] 400`summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.[1] 342
[1] 342`summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.[1] 341
[1] 341`summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.[1] 341処理終わり!
ということで関数とwhileなどの繰り返し処理を利用すると、すっきりと書くことができます。
最後に、ここで処理したデータを利用して、最初に求めようとしていた、薬A、B、Cの投与期間の最小値、最大値、平均値をもとめてみましょう。
dat_n3# A tibble: 341 × 4
id med start end
<dbl> <chr> <date> <date>
1 1 A 2020-04-14 2020-05-07
2 1 B 2020-04-01 2020-04-09
3 2 A 2020-05-12 2020-06-30
4 3 A 2020-05-31 2020-08-01
5 4 B 2020-04-03 2020-06-04
6 5 A 2020-04-26 2020-07-09
7 5 B 2020-04-15 2020-05-10
8 6 A 2020-05-16 2020-06-14
9 6 B 2020-05-02 2020-05-07
10 7 C 2020-05-28 2020-06-27
# ℹ 331 more rowsdat_n3 %>%
group_by(med) %>%
summarise(
min_kikan = min(end-start),
max_kikan = max(end-start),
avg_kikan = mean(end-start)
)# A tibble: 3 × 4
med min_kikan max_kikan avg_kikan
<chr> <drtn> <drtn> <drtn>
1 A 3 days 135 days 54.87786 days
2 B 1 days 126 days 58.81308 days
3 C 1 days 130 days 48.01942 daysできました!
いかがでしょうか?
だいぶ複雑でしたが、少し工夫して計算することで日付の幅等の集計を行うことができました。
お疲れさまでした。以上で集計に関する内容はおしまいです。
補足として、intervalを利用しないで同様の処理を行う方法を次の動画で解説しておりますので、よろしければどうぞ。
必要ないという方は次のセクションにお進みください