library(tidyverse)
library(lubridate)
plot_id <- function(.data,tgt_id){
gdat <- .data %>%
filter(id %in% tgt_id) %>%
mutate(row_n = n():1) %>%
mutate(points = map2(start,end, ~{.x:.y})) %>%
select(id,med,row_n,points) %>%
unnest(c(points)) %>%
mutate(points = as_date(points))
ggplot(gdat) +
geom_point(aes(x = points, y = as.factor(row_n), color = med)) +
scale_y_discrete(labels=NULL) +
facet_wrap(~id, scales = "free")
}ここまでの解説では、各IDの薬剤毎ということは加味しておりませんでしたが、ここからはそこも加味して処理を書いていきます。
ご自身で試みてみましたか?
この処理ですが、id列とmed列を含んだ処理を書いてく形ですやってみましょう
dat <- read_csv("data/time.csv")Rows: 400 Columns: 4
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): med
dbl (1): id
date (2): start, end
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.dat2 <- dat %>%
arrange(id,med,start) %>% #ここにmed,startを追加
mutate(interv = interval(start, end)) %>% #同じ
group_by(id, med) %>% #idとmedでグループ化
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_edgealign = int_aligns(interv, lag(interv)),
) %>%
mutate(prev_oa = prev_overlap | prev_edgealign) #グループ化後は同じ
View(dat2)うまく、重複・期間の端が隣接している行をTRUEとふれていますね?
あとは、NAをFALSEで埋めてあげて、
dat3 <- dat2 %>%
replace_na(list(prev_oa = FALSE))
View(dat3)各IDの各処方毎、処方期間の重複をふくめて、IDを振ってあげます。
dat3# A tibble: 400 × 8
# Groups: id, med [326]
id med start end interv prev_overlap
<dbl> <chr> <date> <date> <Interval> <lgl>
1 1 A 2020-04-14 2020-05-07 2020-04-14 UTC--2020-05-07 UTC NA
2 1 B 2020-04-01 2020-04-09 2020-04-01 UTC--2020-04-09 UTC NA
3 2 A 2020-05-12 2020-06-30 2020-05-12 UTC--2020-06-30 UTC NA
4 2 A 2020-05-24 2020-06-17 2020-05-24 UTC--2020-06-17 UTC TRUE
5 3 A 2020-05-31 2020-08-01 2020-05-31 UTC--2020-08-01 UTC NA
6 4 B 2020-04-03 2020-06-04 2020-04-03 UTC--2020-06-04 UTC NA
7 5 A 2020-04-26 2020-07-09 2020-04-26 UTC--2020-07-09 UTC NA
8 5 B 2020-04-15 2020-05-10 2020-04-15 UTC--2020-05-10 UTC NA
9 5 B 2020-04-15 2020-04-28 2020-04-15 UTC--2020-04-28 UTC TRUE
10 6 A 2020-05-16 2020-06-14 2020-05-16 UTC--2020-06-14 UTC NA
# ℹ 390 more rows
# ℹ 2 more variables: prev_edgealign <lgl>, prev_oa <lgl>にかけたグループはまだそのまま残っているので、単純に、mutateするだけでよくて、ここも、処理内容は変わりません
dat4 <- dat3 %>%
mutate(presc_id = cumsum(!prev_oa))
plot_id(dat, c(39,140))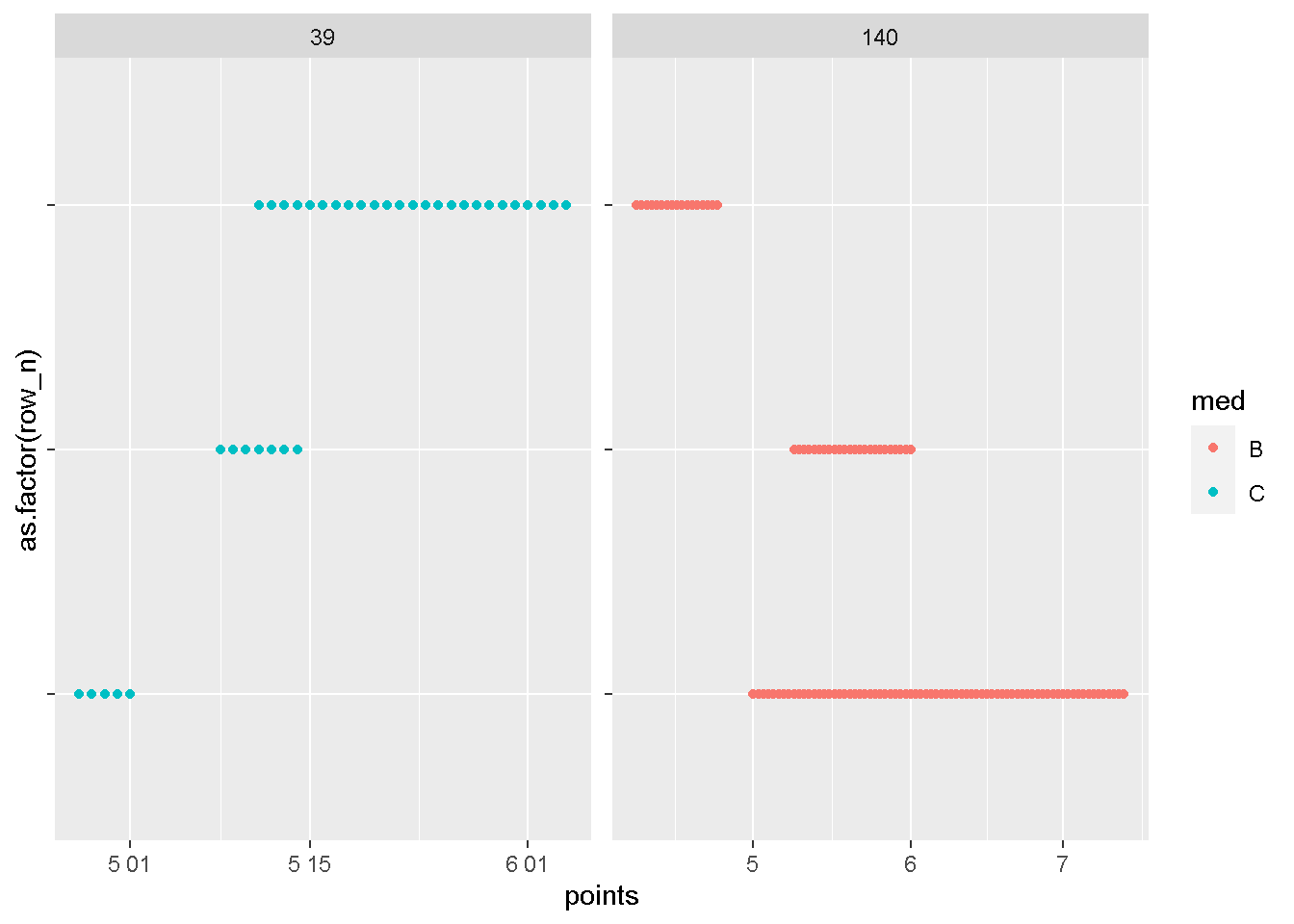
dat4 %>%
filter(id %in% c(39,140)) %>%
select(id,med,start,end,presc_id)# A tibble: 6 × 5
# Groups: id, med [2]
id med start end presc_id
<dbl> <chr> <date> <date> <int>
1 39 C 2020-04-27 2020-05-01 1
2 39 C 2020-05-08 2020-05-14 2
3 39 C 2020-05-11 2020-06-04 2
4 140 B 2020-04-08 2020-04-24 1
5 140 B 2020-05-01 2020-07-13 2
6 140 B 2020-05-09 2020-06-01 2いかがでしょうか?うまく、「重複毎」に、presc_idが振られていますね?
後は、
dat5 <- dat4 %>%
group_by(id, med, presc_id) %>% #グループを作り直し。id,medを追加
summarise(start = min(start), end = max(end)) # 同じ`summarise()` has grouped output by 'id', 'med'. You can override using the
`.groups` argument.うまくいきました。
尚、ここで、ちょっとだけ注意が必要なのが、summarise実行時にでているメッセージです
summarise() has grouped output by ‘id’, ‘med’.You can override using the .groups argument.
とあるのですが、これは、summarise実行ででてきた結果に「id med」の二つのグループが残っているというメッセージです。
実際、
dat5# A tibble: 346 × 5
# Groups: id, med [326]
id med presc_id start end
<dbl> <chr> <int> <date> <date>
1 1 A 1 2020-04-14 2020-05-07
2 1 B 1 2020-04-01 2020-04-09
3 2 A 1 2020-05-12 2020-06-30
4 3 A 1 2020-05-31 2020-08-01
5 4 B 1 2020-04-03 2020-06-04
6 5 A 1 2020-04-26 2020-07-09
7 5 B 1 2020-04-15 2020-05-10
8 6 A 1 2020-05-16 2020-06-14
9 6 B 1 2020-05-02 2020-05-07
10 7 C 1 2020-05-28 2020-06-27
# ℹ 336 more rowsで確認すると確かにグループが残存しています。sumariseでは最後のグループだけが解消されるイメージです。このグループを消したい場合は、
dat5 %>% ungroup()# A tibble: 346 × 5
id med presc_id start end
<dbl> <chr> <int> <date> <date>
1 1 A 1 2020-04-14 2020-05-07
2 1 B 1 2020-04-01 2020-04-09
3 2 A 1 2020-05-12 2020-06-30
4 3 A 1 2020-05-31 2020-08-01
5 4 B 1 2020-04-03 2020-06-04
6 5 A 1 2020-04-26 2020-07-09
7 5 B 1 2020-04-15 2020-05-10
8 6 A 1 2020-05-16 2020-06-14
9 6 B 1 2020-05-02 2020-05-07
10 7 C 1 2020-05-28 2020-06-27
# ℹ 336 more rowsとグループを消す処理を意図的に入れてあげるか、
dat4 %>%
group_by(id, med, presc_id) %>%
summarise(start = min(start), end = max(end), .groups = "drop")# A tibble: 346 × 5
id med presc_id start end
<dbl> <chr> <int> <date> <date>
1 1 A 1 2020-04-14 2020-05-07
2 1 B 1 2020-04-01 2020-04-09
3 2 A 1 2020-05-12 2020-06-30
4 3 A 1 2020-05-31 2020-08-01
5 4 B 1 2020-04-03 2020-06-04
6 5 A 1 2020-04-26 2020-07-09
7 5 B 1 2020-04-15 2020-05-10
8 6 A 1 2020-05-16 2020-06-14
9 6 B 1 2020-05-02 2020-05-07
10 7 C 1 2020-05-28 2020-06-27
# ℹ 336 more rowsと、summariseの時点で.group引数に”drop”を与えるとsummarise時点でgroupが消えます。こちらの方法、本コースを作成している時点では、実験的な機能なので、将来使えなくなる可能性もあるのでその点ご留意ください。このコースでは、ungroupを主に利用します。それで、
dat_fin <- dat5 %>%
ungroup() %>%
arrange(id,med,start) #並び替えておきます。これで完成です。
可視化して確認しておきましょう
compare_plot <- function(.data1, .data2, tgt_id){
pre_graph <- plot_id(.data1, tgt_id)
post_graph <- plot_id(.data2, tgt_id)
cowplot::plot_grid(pre_graph, post_graph, nrow=2)
}
compare_plot(dat,dat_fin,c(2,5,10))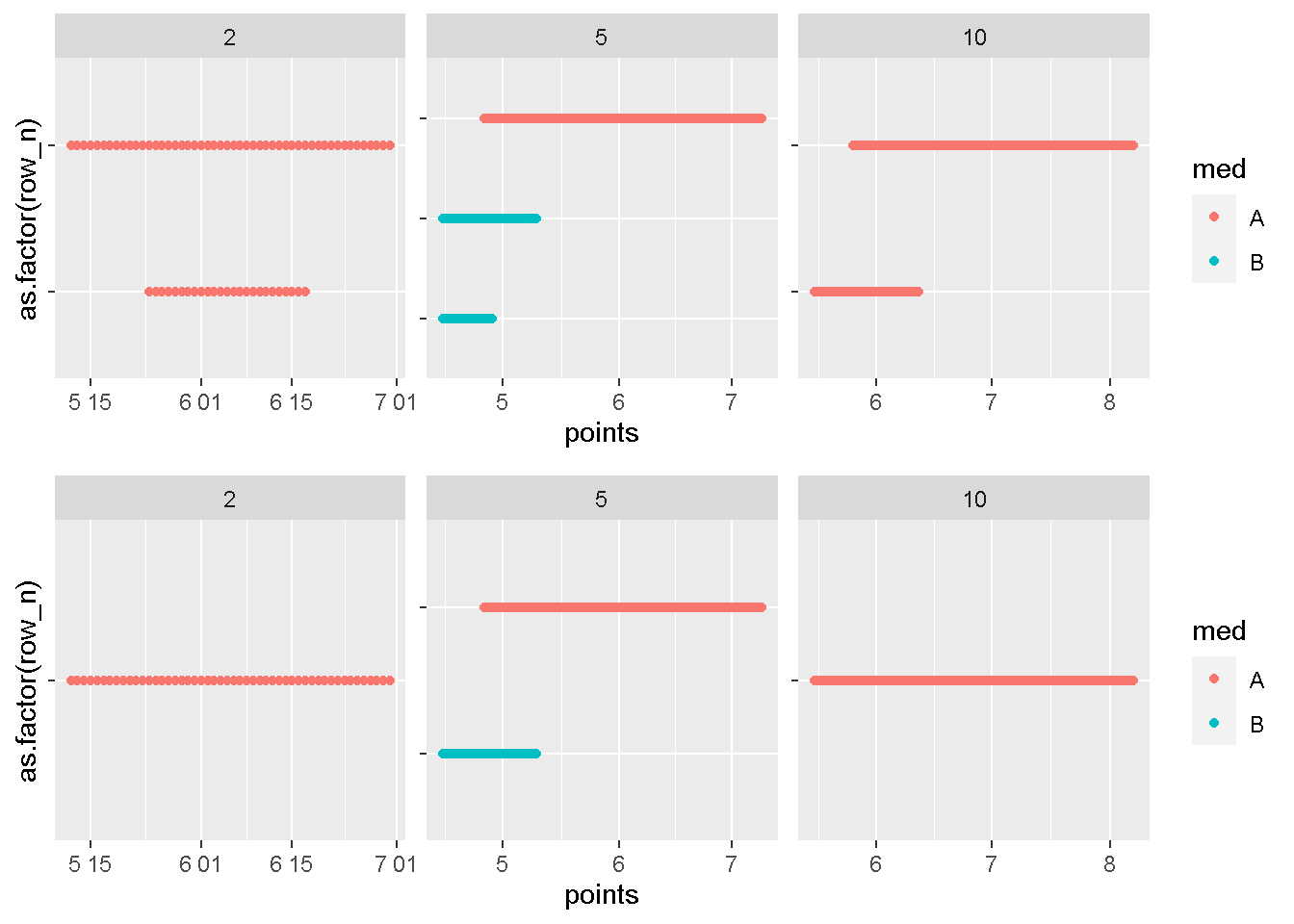
上のグラフがもとのもので、下のグラフが処理後のものですうまく期間が一つにID、薬毎にまとまっていますね?
ただし、今回の処理、一つ上のものとの比較だけをしているため、
compare_plot(dat,dat_fin,c(39,93,140))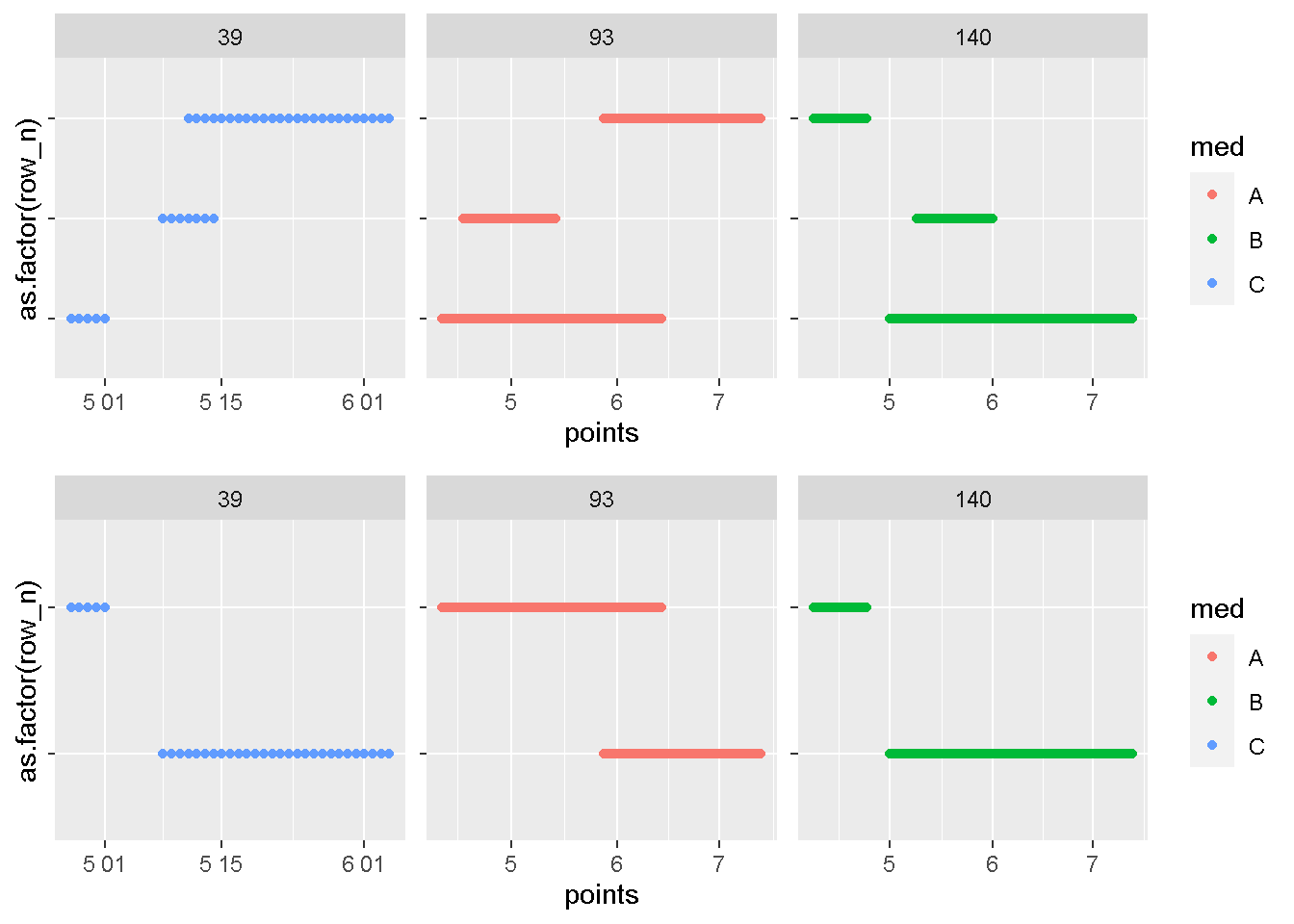
id 93番が想定した結果と少し違います。これ、なぜかわかりますか?
少し考えてみてください。次の動画で解説していきます。