library(tidyverse)
dat <- read_csv(
file = "data/mc360000.csv",
locale=locale(encoding="shift-jis"),
skip = 3,
col_names = FALSE
)
row1 <- dat %>% slice(1) %>% as_vector()
row2 <- dat %>% slice(2) %>% as_vector()
row3 <- dat %>% slice(3) %>% as_vector()
dat_colname <- tibble(
r1 = row1,
r2 = row2,
r3 = row3
)
dat_colname <- dat_colname %>% fill(r1,r2,r3,.direction="down")
dat_colname2 <- dat_colname %>% unite(col = "coln", r1, r2, r3,sep="_", remove=FALSE)
vec_coln <- dat_colname2 %>% pull(coln)
dat2 <- dat %>% setNames(vec_coln)
dat3 <- dat2 %>% slice(-(1:4))
dat4 <- dat3 %>% rename(cause = NA_NA_NA)もう少しです。cause列、[X数字 説明]という形になっているため、これは二つに分けておきます。
区切られている文字をしらべてきます
str_view_all(dat4$cause[1:3]," ")Warning: `str_view()` was deprecated in stringr 1.5.0.
ℹ Please use `str_view_all()` instead.[1] │ X60{\u3000\u3000\u3000}非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬
[2] │ X61{\u3000\u3000\u3000}抗てんかん薬,鎮静・催眠薬,パーキンソン病治療薬及び向精神薬
[3] │ X62{\u3000\u3000\u3000}麻薬及び精神変容薬[幻覚発現薬]str_view_all(dat4$cause[1:3]," ")[1] │ X60<{\u3000}><{\u3000}><{\u3000}>非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬
[2] │ X61<{\u3000}><{\u3000}><{\u3000}>抗てんかん薬,鎮静・催眠薬,パーキンソン病治療薬及び向精神薬
[3] │ X62<{\u3000}><{\u3000}><{\u3000}>麻薬及び精神変容薬[幻覚発現薬]どうやら全角文字のようなので、
dat5 <- dat4 %>%
separate(cause, c("id","name"), sep=" +")
View(dat5)準備がととのいました!
dat6 <- dat5 %>%
pivot_longer(
cols = !c(id,name),
names_to = c("sex","type","year"),
values_to = "val",
names_sep = "_"
)
View(dat6)ほとんどtidyです
str(dat6)tibble [1,500 × 6] (S3: tbl_df/tbl/data.frame)
$ id : chr [1:1500] "X60" "X60" "X60" "X60" ...
$ name: chr [1:1500] "非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬" "非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬" "非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬" "非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬" ...
$ sex : chr [1:1500] "総数" "総数" "総数" "総数" ...
$ type: chr [1:1500] "死亡数" "死亡数" "死亡数" "死亡数" ...
$ year: chr [1:1500] "1995" "2000" "2005" "2010" ...
$ val : chr [1:1500] "8" "10" "7" "8" ...yearとvalを数値型に変換しておきましょう。また、valは未測定は「-」で記入されているのでそこは欠損値にしておきます(わざわざ変換しなくても、NAに-は勝手におきかわりますが、警告がでるのも気持ち悪いので手動で変換しておきましょう。)
dat7 <- dat6 %>%
mutate(year = as.numeric(year),
val = val %>% na_if("-") %>% as.numeric())
View(dat7)総数がsexに含まれているは気持ち悪いのでこれも消しておきましょう。
dat8 <- dat7 %>%
filter(sex != "総数")以上、ここまでの処理で、
View(dat)が、
View(dat7)になった結果、
gdat <- dat8 %>%
filter(type == "死亡数") %>%
filter(id == "X60")
title <- gdat$name[1]
ggplot(gdat) +
geom_col(aes(x = year, y = val, fill = sex)) +
facet_wrap(~sex) +
labs(title = title)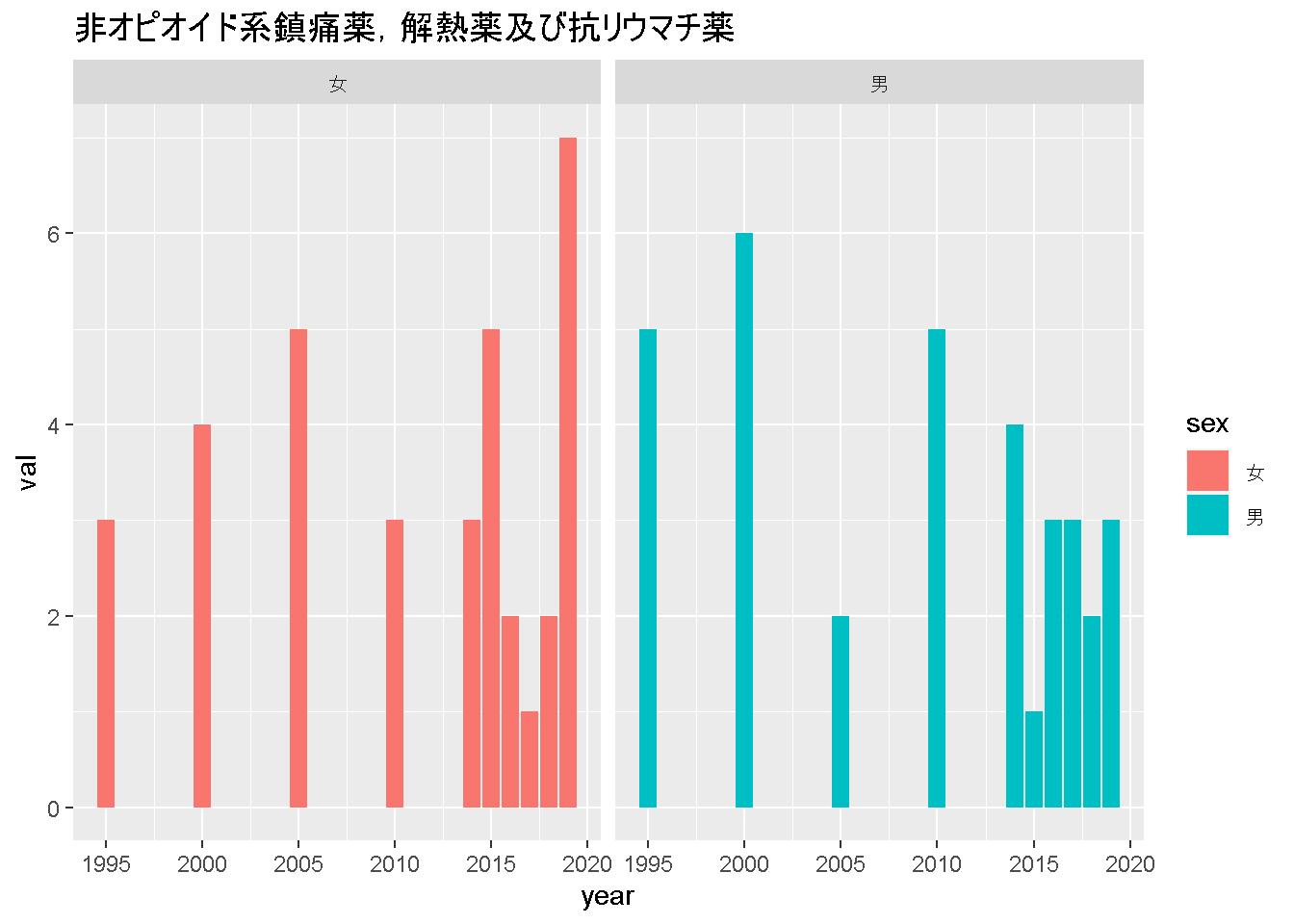
と、こんな感じでグラフを簡単に描画できました。
他にもdistinct関数を利用すると
dat8 %>%
select(id, name) %>%
distinct()# A tibble: 25 × 2
id name
<chr> <chr>
1 X60 非オピオイド系鎮痛薬,解熱薬及び抗リウマチ薬
2 X61 抗てんかん薬,鎮静・催眠薬,パーキンソン病治療薬及び向精神薬
3 X62 麻薬及び精神変容薬[幻覚発現薬]
4 X63 自律神経系に作用するその他の薬物
5 X64 その他及び詳細不明の薬物,薬剤及び生物学的製剤
6 X65 アルコール
7 X66 有機溶剤及びハロゲン化炭化水素類及びそれらの蒸気
8 X67 その他のガス及び蒸気
9 X68 農薬
10 X69 その他及び詳細不明の化学物質及び有害物質
# ℹ 15 more rowsこんな感じで集計することができます。
薬剤関係でX60,X61,X62,X63の4つのidを絞り込んで描画するのであれば、
gdat <- dat8 %>%
filter(type == "死亡数") %>%
filter(id %in% c("X60","X61","X62","X63"))
ggplot(gdat) +
geom_col(aes(x = year, y = val, fill = sex)) +
facet_wrap(~name)Warning: Removed 23 rows containing missing values (`position_stack()`).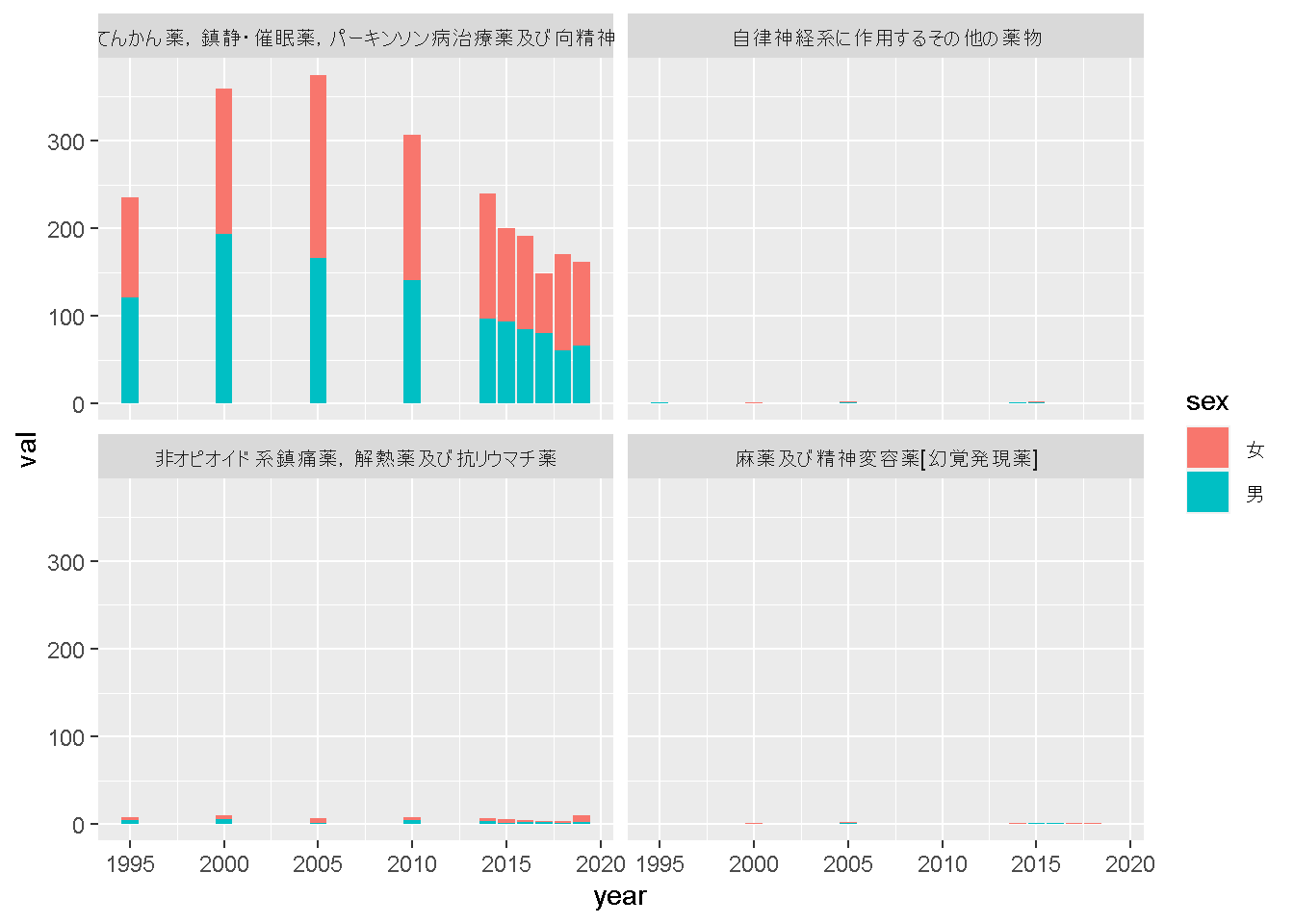
というグラフを作成することもできます。
尚、%in% という表記をはじめて利用しましたが、
c(1,2,3, 1, 2, 3, 4, 5, 1) %in% c(2, 4)[1] FALSE TRUE FALSE FALSE TRUE FALSE TRUE FALSE FALSEこんな感じで、左側のベクトルに対して、右側のベクトルに含まれているかそうでないかという条件でロジカルベクトルを作成することができます。
これを、filterの中で利用すれば、複数の条件で絞り込むことが簡単にできます。
次はここで学んだ智識を使って、食中毒データの処理を行いましょう。