library(tidyverse)
library(gtsummary)Warning: package 'gtsummary' was built under R version 4.3.2library(tidyverse)
library(gtsummary)Warning: package 'gtsummary' was built under R version 4.3.2課題3: 上司から、BMIが30以上の人、BMIが16未満の人、あるいは血圧が160/100どちらかが超えている人を抽出して、個々のグラフを作成して、それを利用して産業看護職として健康相談をおこなうように指示がありました。 これまで、エクセルで条件に当てはまる人を抽出して、グラフと表を作成して、Wordファイルに貼り付けたうえで、コメントを書いていました。 Wordファイルを作成するところまでをRでやってみましょう。今回は、面談時に提示するだけなので、特に凝ったレイアウトは必要ありません。
最後の課題です。
まずは、条件に適合する人を抽出しましょう。
dat <- read_csv("kadai/data/data.csv")Rows: 2460 Columns: 6
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (6): wpid, id, yr, bmi, sbp, dbp
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.filtdat <- dat %>%
filter(bmi >= 30 | bmi < 16 | sbp >= 160 | dbp >= 100) 35行が該当しました。(注:このデータはランダムに作成した架空のデータです。)
対象者、複数回該当しているケースもあるので、distinctで重複を消しておきましょう。
filtdat %>%
distinct(wpid, id)# A tibble: 20 × 2
wpid id
<dbl> <dbl>
1 4 1
2 4 5
3 4 6
4 4 8
5 4 9
6 4 10
7 4 11
8 4 14
9 4 16
10 4 18
11 4 20
12 4 25
13 4 28
14 4 31
15 4 32
16 4 33
17 4 34
18 4 36
19 4 37
20 4 4020名分、つまり、20個のWordファイルの作成が必要という形です。
一人分のWordファイルに出力するグラフと表を作成してみます。
adat <- dat %>%
filter(wpid==4 & id == 1) %>%
mutate(yr = as.factor(yr))
ggplot(adat) +
geom_line(aes(x = yr, y = bmi, group=1)) +
geom_point(aes(x = yr, y = bmi)) +
theme_classic() +
labs(x = "年度", y = "BMI")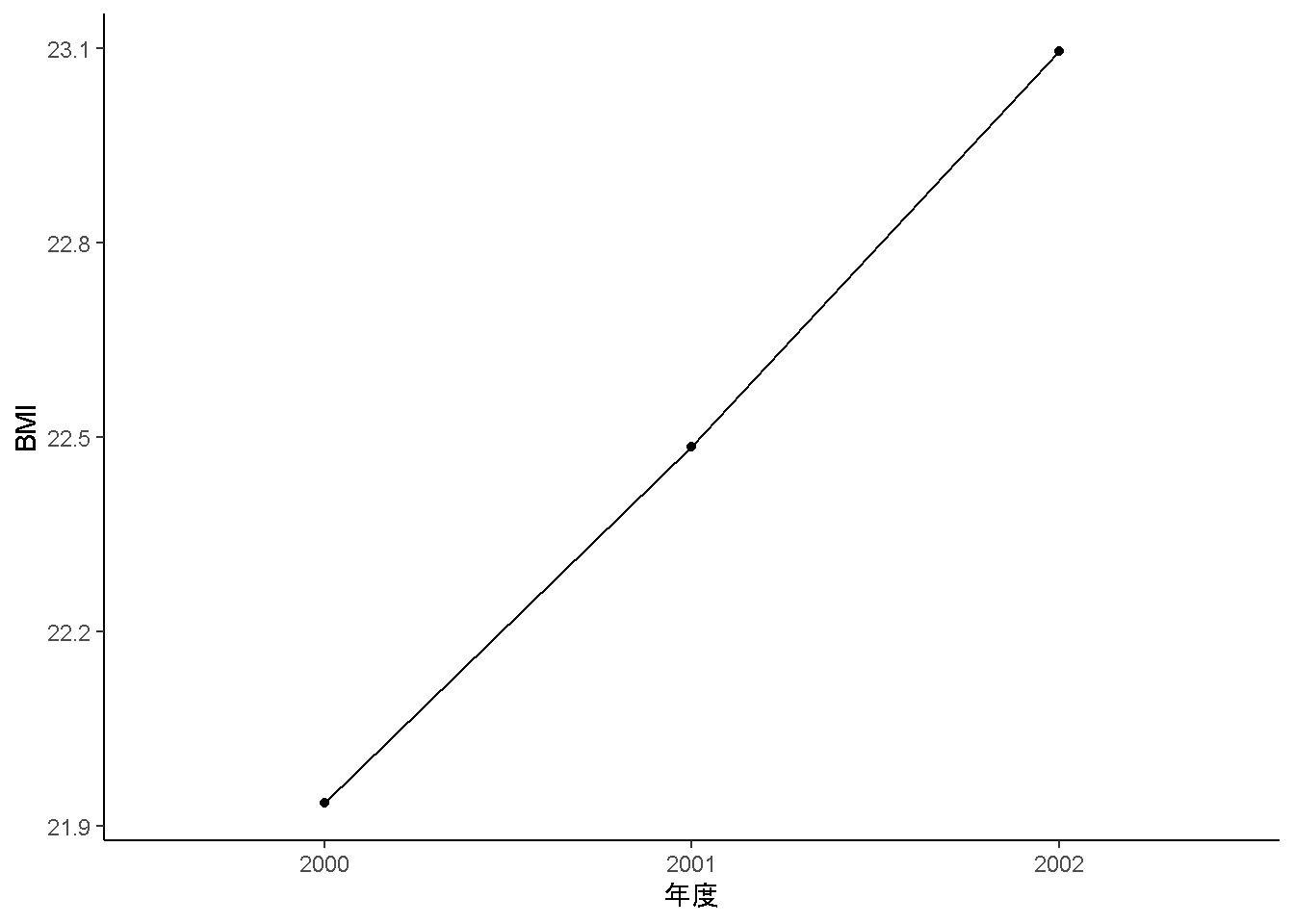
geomを重ねる話、本コースでは行っていませんが、
このように重ねることも可能です(詳しくは中級者向けのグラフ描画コースで解説しています)
他にも、
adat %>%
pivot_longer(c(sbp,dbp)) %>%
ggplot(aes(x = yr, y = value, shape=name, group=name)) +
geom_point() +
geom_line() +
geom_hline(yintercept = c(80,120), linetype="dotted", color="black") +
geom_hline(yintercept = c(90,140), linetype="dotted", color="orange") +
geom_hline(yintercept = c(100,160), linetype="dotted", color="red") +
scale_shape_discrete(name="血圧")+
labs(x = "年度", y = "値", title = "血圧の推移") +
theme_classic()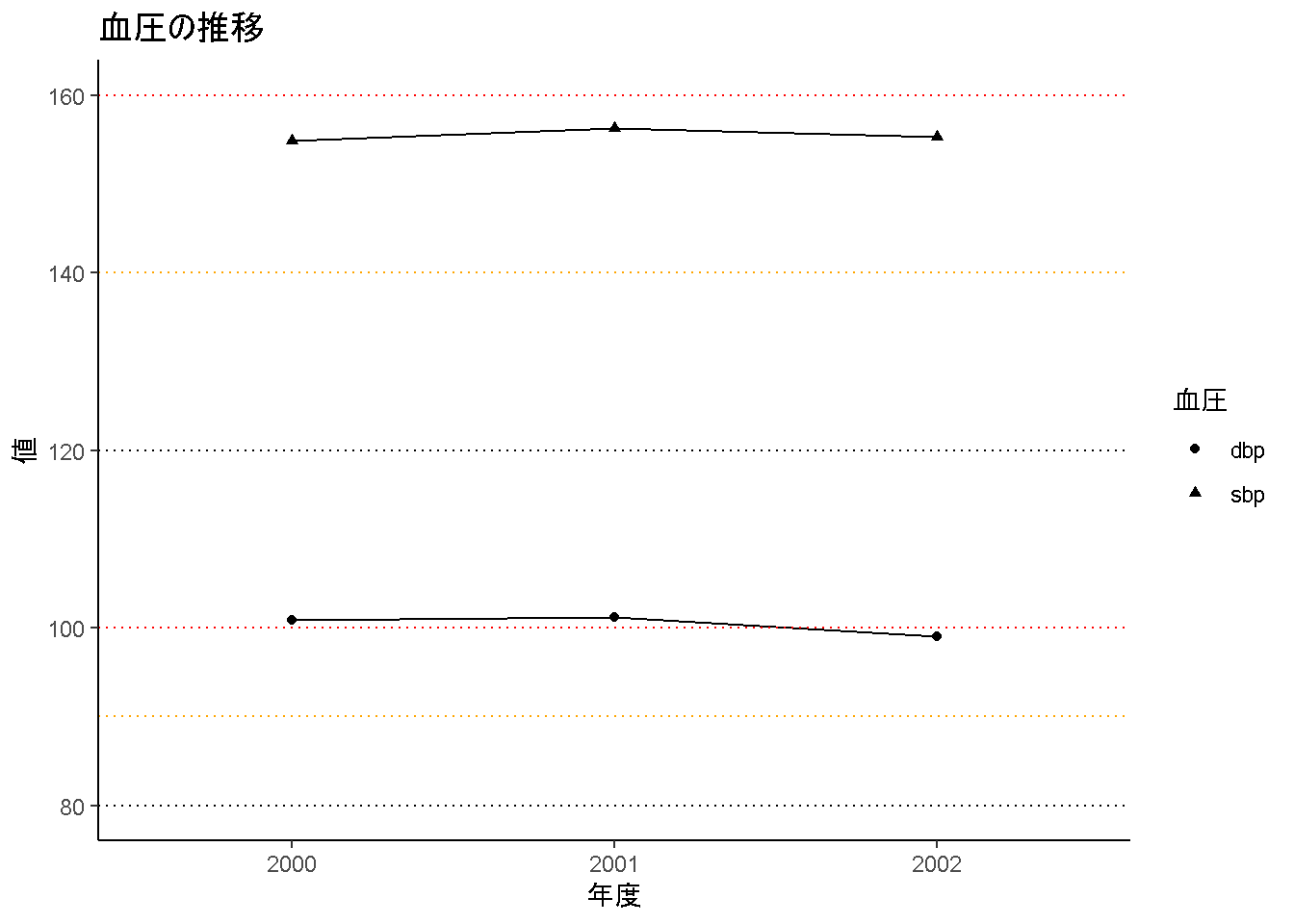
こんな感じのグラフを作成することもできます。
今回は、この2種類のグラフと次の表
adat %>%
select(yr,bmi,sbp,dbp) %>%
mutate(across(c(bmi,sbp,dbp),~{format(signif(.,3))})) %>%
rename(`年度` = yr, `BMI` = bmi, `収縮期血圧` = sbp, `拡張期血圧` = dbp)# A tibble: 3 × 4
年度 BMI 収縮期血圧 拡張期血圧
<fct> <chr> <chr> <chr>
1 2000 21.9 155 "101"
2 2001 22.5 156 "101"
3 2002 23.1 155 " 99" を含んだWordファイルを全員分作ることとします。
ここで20名分のWordファイルを作るのに、Rmdファイルは1つで十分です。
パラメーターレポートという、Rmarkdownの内容を簡単に置き換えられる仕組みを使って、20名分のレポートを作成してみましょう。
まず、kadai3.Rmdを開いて中身を確認してみてください。
knitした結果がwordファイルになっていますね?
このkadai3.Rmdをパラメーターレポートにしてあげます。
kadai3param.Rmdを開いてみてください。
kadai3param.Rmdレポートを外から設定を変更して実行してみましょう。
rmarkdown::render(input = "kadai/kadai3param.Rmd",
output_dir = "kadai/kojinreport",
params = list(targetwpid = 1, targetid = 1),
output_file = "test.docx")どうでしょう?うまく実行できましたね?
後は、これを繰り返し処理するスクリプトを書いて実行すると、うまくいきます。
繰り返し処理については、このコースでは解説していませんでしたが、ここで簡単に解説しておきます。
繰り返し処理は、forループというものを利用します。
for(i in 1:10){
print(i)
}[1] 1
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10最初はちょっとややこしいかもしれませんが、このようにforで始まる構文で、{}の中身に繰り返したい処理を書いてあげることで、ジョジョに数字を変えながら{}の中身を実行することができます。
今回、
dat <- read_csv("kadai/data/data.csv")Rows: 2460 Columns: 6
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (6): wpid, id, yr, bmi, sbp, dbp
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.filtdat <- dat %>%
filter(bmi >= 30 | bmi < 16 | sbp >= 160 | dbp >= 100)
filtdat <- filtdat %>%
distinct(wpid, id)このfiltdatの1行1行の組み合わせでwordファイルを作りたいので、
そうなるようにforループを作成してみましょう。
for(i in 1:nrow(filtdat)){
tgtwpid <- filtdat$wpid[i]
tgtid <- filtdat$id[i]
print(str_c("wpid:",tgtwpid, "/", "id:",tgtid))
}[1] "wpid:4/id:1"
[1] "wpid:4/id:5"
[1] "wpid:4/id:6"
[1] "wpid:4/id:8"
[1] "wpid:4/id:9"
[1] "wpid:4/id:10"
[1] "wpid:4/id:11"
[1] "wpid:4/id:14"
[1] "wpid:4/id:16"
[1] "wpid:4/id:18"
[1] "wpid:4/id:20"
[1] "wpid:4/id:25"
[1] "wpid:4/id:28"
[1] "wpid:4/id:31"
[1] "wpid:4/id:32"
[1] "wpid:4/id:33"
[1] "wpid:4/id:34"
[1] "wpid:4/id:36"
[1] "wpid:4/id:37"
[1] "wpid:4/id:40"tgtwpidとtgtidという変数にそれぞれ、i行目の値が含まれていますね?
これを利用して、先ほどのrmarkdown::renderを実行してあげましょう。
for(i in 1:nrow(filtdat)){
tgtwpid <- filtdat$wpid[i]
tgtid <- filtdat$id[i]
filename <- str_c("repoort_",tgtwpid,"_",tgtid,".docx")
print(str_c("wpid:",tgtwpid, "/", "id:",tgtid))
rmarkdown::render(input = "kadai/kadai3param.Rmd",
output_dir = "kadai/kojinreport",
params = list(targetwpid = tgtwpid, targetid = tgtid),
output_file = filename)
}このforループでkadai/kojineportの中には、20個のwordファイルができています。
list.files("kadai/kojinreport/",pattern="docx")それぞれの中身は、ファイル名にあるように、各々のidのデータが反映されたグラフと表になっているはずです。