library(tidyverse)
library(readxl)ここでは、data/fill2.csvを利用します1つ前の練習問題で作成していない場合は、次のコードを実行してください。
read_excel("data/fill.xlsx") %>%
rename(`cause1` = `病因物質`,
`cause2` = `...2`) %>%
mutate(
cause1 = str_replace_all(cause1,"\\s",""),
cause2 = str_replace_all(cause2,"\\s","")
) %>%
fill(cause1) %>%
filter(!is.na(cause2)) %>%
write_csv("data/fill2.csv")New names:
• `` -> `...2`練習問題1:
“data/fill2.csv”をインポートしてください。
練習問題2:
cause1, cause2, 事件_1, 患者_1, 死者_1,を選択してください。
練習問題3:
事件_1、患者_1、死者_1をそれぞれcase_1, pt_1, death_1へと名前を変換してください。
練習問題4:
case_1, pt_1, death_1の値で「-」となっている値をNA、欠損値に置き換えてください
練習問題5:
表の欠損値を0に置き換えてください。
練習問題6:
case_1, pt_1, death_1 を数字に変換して、その値を利用してx軸をcause2, y軸をcase_1として棒グラフを描画してください。
練習問題1:
“data/fill2.csv”をインポートしてください。
dat1 <- read_csv("data/fill2.csv")Rows: 24 Columns: 38
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (38): cause1, cause2, 事件_1, 患者_1, 死者_1, 事件_2, 患者_2, 死者_2, 事件_3, 患者_3, 死者...
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.練習問題2:
cause1, cause2, 事件_1, 患者_1, 死者_1,を選択してください。
dat2 <- dat1 %>%
select(cause1, cause2, ends_with("_1"))練習問題3:
事件_1、患者_1、死者_1をそれぞれcase_1, pt_1, death_1へと名前を変換してください。
dat3 <- dat2 %>%
rename(case_1 = `事件_1`,
pt_1 = `患者_1`,
death_1 = `死者_1`)
dat3# A tibble: 24 × 5
cause1 cause2 case_1 pt_1 death_1
<chr> <chr> <chr> <chr> <chr>
1 細菌 サルモネラ属菌 1 95 -
2 細菌 ぶどう球菌 1 28 -
3 細菌 ボツリヌス菌 - - -
4 細菌 腸炎ビブリオ - - -
5 細菌 腸管出血性大腸菌(VT産生) - - -
6 細菌 その他の病原大腸菌 - - -
7 細菌 ウエルシュ菌 1 10 -
8 細菌 セレウス菌 - - -
9 細菌 エルシニア・エンテロコリチカ - - -
10 細菌 カンピロバクター・ジェジュニ/コリ 5 21 -
# ℹ 14 more rows練習問題4:
case_1, pt_1, death_1の値で「-」となっている値をNA、欠損値に置き換えてください
dat4 <- dat3 %>%
mutate(
case_1 = na_if(case_1,"-"),
pt_1 = na_if(pt_1,"-"),
death_1 = na_if(death_1,"-")
)
dat4# A tibble: 24 × 5
cause1 cause2 case_1 pt_1 death_1
<chr> <chr> <chr> <chr> <chr>
1 細菌 サルモネラ属菌 1 95 <NA>
2 細菌 ぶどう球菌 1 28 <NA>
3 細菌 ボツリヌス菌 <NA> <NA> <NA>
4 細菌 腸炎ビブリオ <NA> <NA> <NA>
5 細菌 腸管出血性大腸菌(VT産生) <NA> <NA> <NA>
6 細菌 その他の病原大腸菌 <NA> <NA> <NA>
7 細菌 ウエルシュ菌 1 10 <NA>
8 細菌 セレウス菌 <NA> <NA> <NA>
9 細菌 エルシニア・エンテロコリチカ <NA> <NA> <NA>
10 細菌 カンピロバクター・ジェジュニ/コリ 5 21 <NA>
# ℹ 14 more rows練習問題5:
表の欠損値を0に置き換えてください。
dat5 <- dat4 %>%
mutate(
case_1 = as.numeric(case_1),
pt_1 = as.numeric(pt_1),
death_1 = as.numeric(death_1),
) %>%
replace_na(list(case_1 = 0, pt_1 = 0, death_1 = 0))
dat5# A tibble: 24 × 5
cause1 cause2 case_1 pt_1 death_1
<chr> <chr> <dbl> <dbl> <dbl>
1 細菌 サルモネラ属菌 1 95 0
2 細菌 ぶどう球菌 1 28 0
3 細菌 ボツリヌス菌 0 0 0
4 細菌 腸炎ビブリオ 0 0 0
5 細菌 腸管出血性大腸菌(VT産生) 0 0 0
6 細菌 その他の病原大腸菌 0 0 0
7 細菌 ウエルシュ菌 1 10 0
8 細菌 セレウス菌 0 0 0
9 細菌 エルシニア・エンテロコリチカ 0 0 0
10 細菌 カンピロバクター・ジェジュニ/コリ 5 21 0
# ℹ 14 more rows練習問題6:
case_1, pt_1, death_1 を数字に変換して、その値を利用してx軸をcause2, y軸をcase_1として棒グラフを描画してください。
ggplot(dat5) +
geom_col(aes(x = cause2, y = case_1)) +
scale_x_discrete(guide = guide_axis(n.dodge = 6))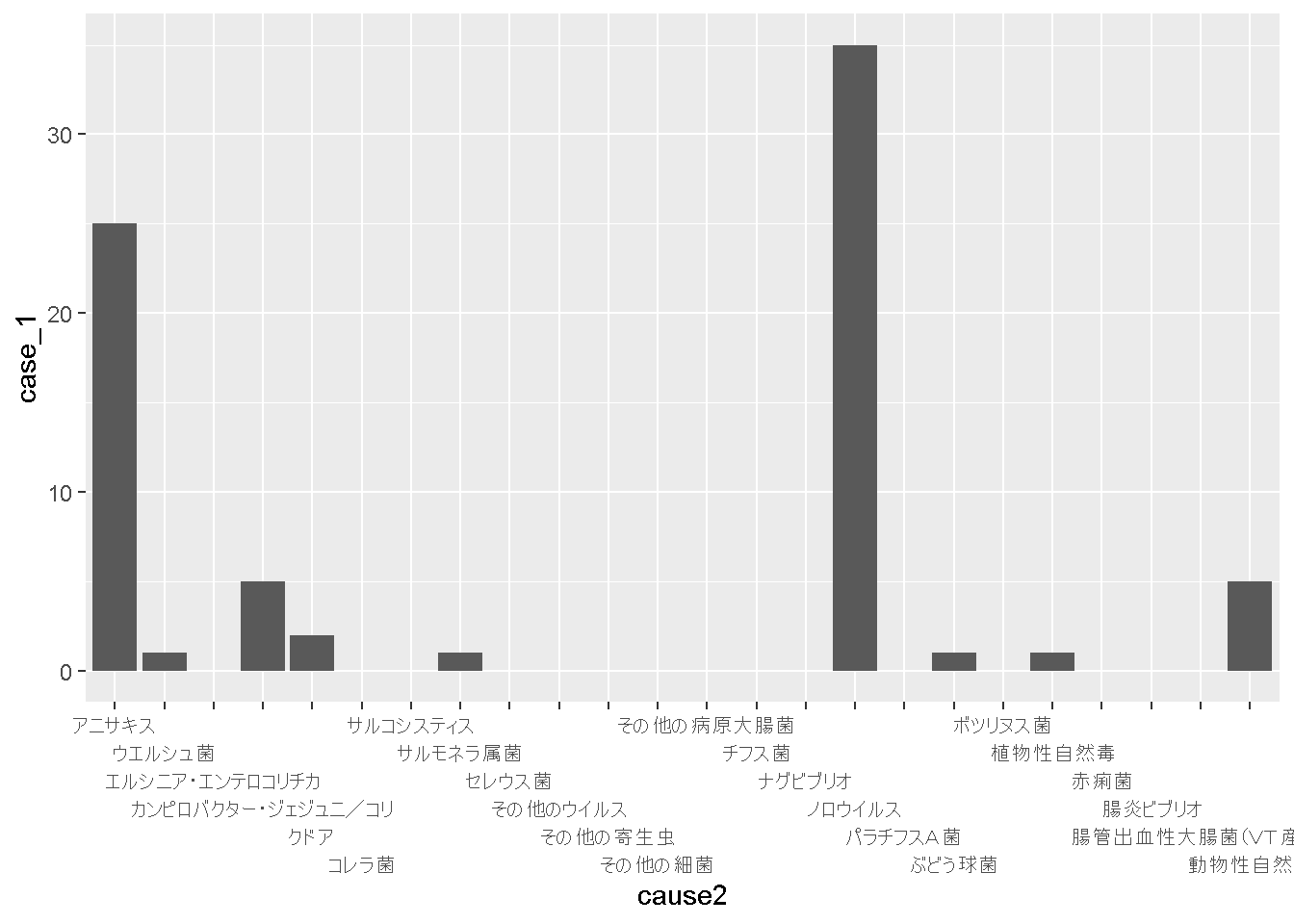
お疲れさまでした!