library(tidyverse)
library(lubridate)
dat <- read_csv("data/time.csv")まず、このデータ、選んだIDを図示してみます。
(解説のための図なので、スクリプトの内容は解説しません)
plot_id <- function(.data,tgt_id){
gdat <- .data %>%
filter(id %in% tgt_id) %>%
mutate(row_n = n():1) %>%
mutate(points = map2(start,end, ~{.x:.y})) %>%
select(id,med,row_n,points) %>%
unnest(c(points)) %>%
mutate(points = as_date(points))
ggplot(gdat) +
geom_point(aes(x = points, y = as.factor(row_n), color = med)) +
scale_y_discrete(labels=NULL) +
facet_wrap(~id, scales = "free")
}このplot_id関数、
plot_id(dat,c(1,15,20))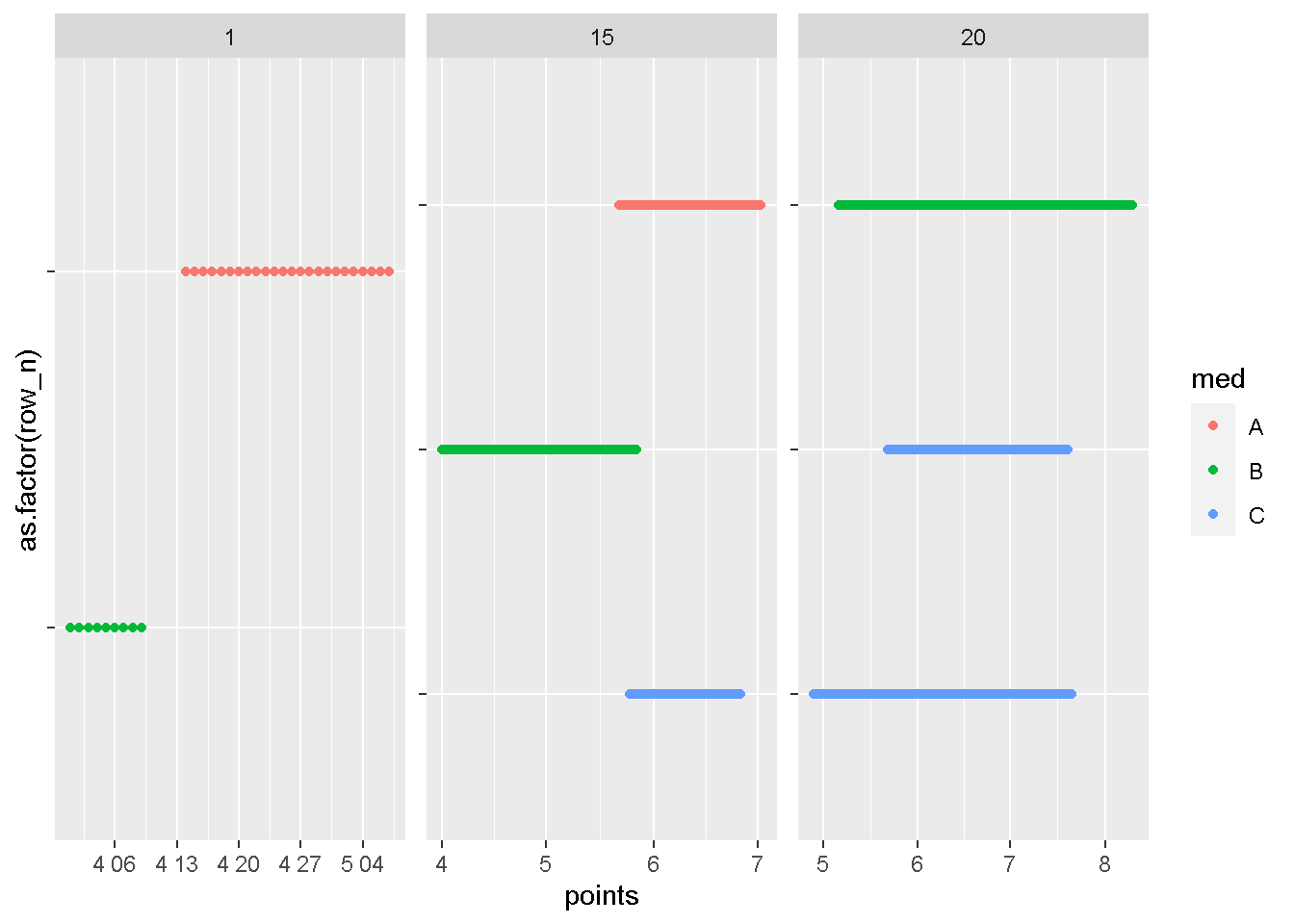
こんな感じで、x軸に日付、y軸に行として、点で処方期間を描画してくれています。
dat %>% filter(id ==20)# A tibble: 3 × 4
id med start end
<dbl> <chr> <date> <date>
1 20 B 2020-05-06 2020-08-10
2 20 C 2020-05-22 2020-07-20
3 20 C 2020-04-28 2020-07-21ここで、課題としては、例えばid20番の青色の点、薬Cの投与期間が重複しているため、これを1つの期間としてまとめたいという形です。
データで、2行より多く行数があるidを抜き出しておきましょう。
id_with_gt2r <- dat %>%
count(id) %>%
filter(n > 2) %>%
pull(id)
id_with_gt2r [1] 5 13 15 16 20 22 23 29 35 36 39 40 45 48 49 57 58 59 62
[20] 66 67 68 75 77 80 81 82 85 86 89 92 93 99 104 108 113 119 125
[39] 128 131 132 140 141 146 147 151 152 159 168 170 173 174 184 187 188 189 192
[58] 194 195 196 200これは
dat %>% count(id)# A tibble: 200 × 2
id n
<dbl> <int>
1 1 2
2 2 2
3 3 1
4 4 1
5 5 3
6 6 2
7 7 1
8 8 1
9 9 1
10 10 2
# ℹ 190 more rowsで、idの数をそれぞれ数えて、nが2より大きいものだけにしぼりこんで、pull関数でベクトルとしてidを抜き出しています。実際にどのようなデータがあるか確認しておきます
plot_id(dat,id_with_gt2r[1:9])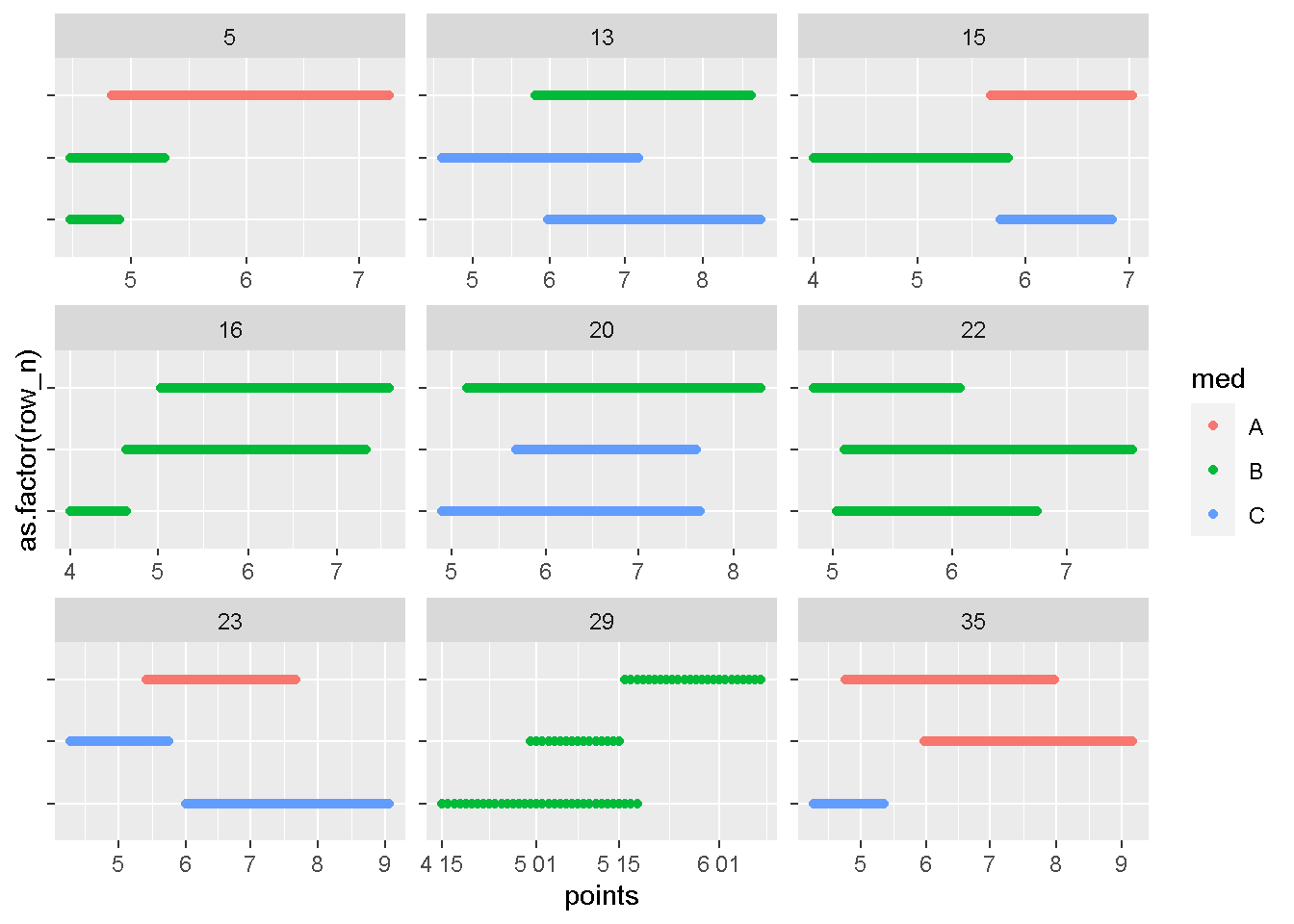
plot_id(dat,id_with_gt2r[10:18]) 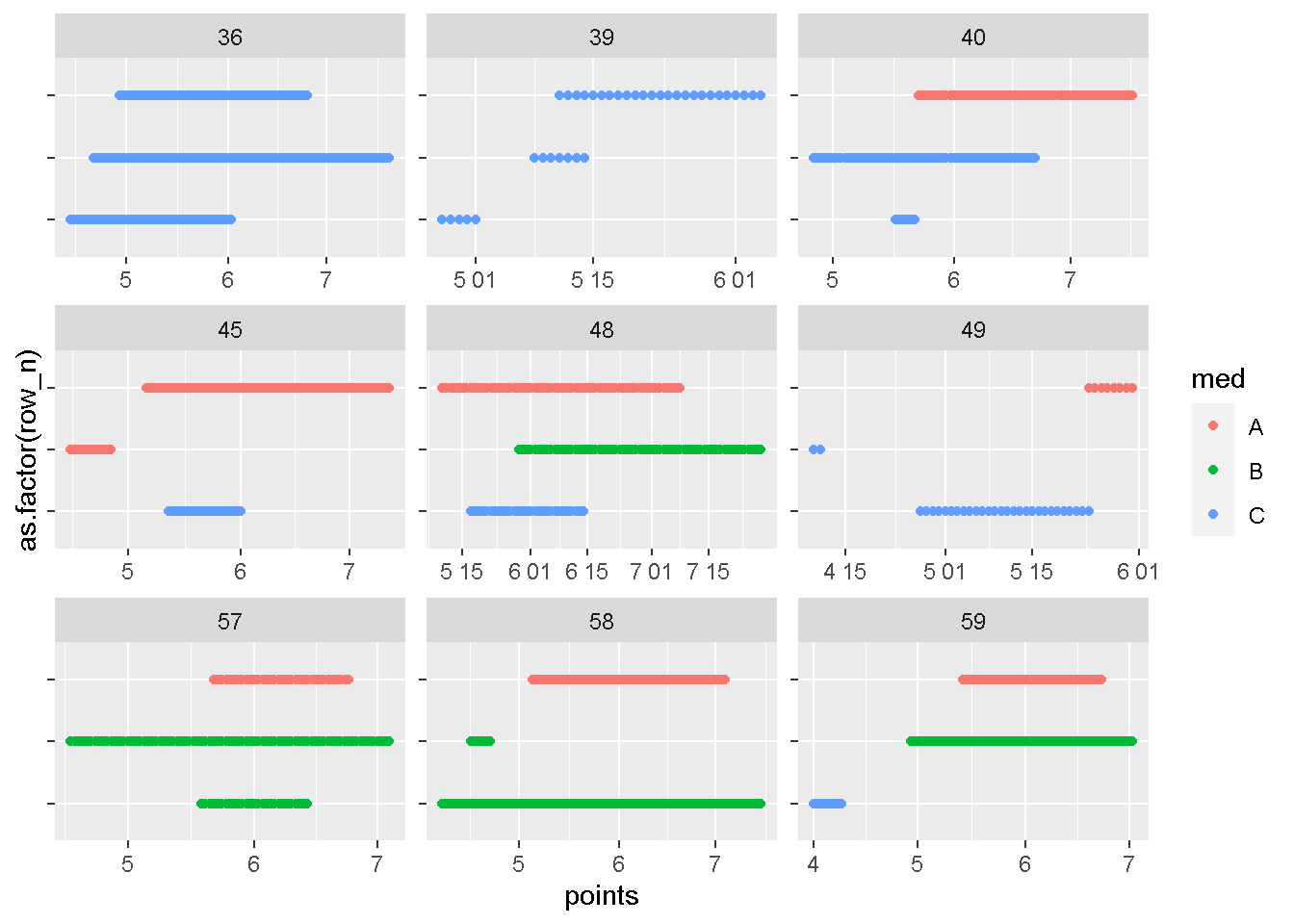
plot_id(dat,id_with_gt2r[19:27]) 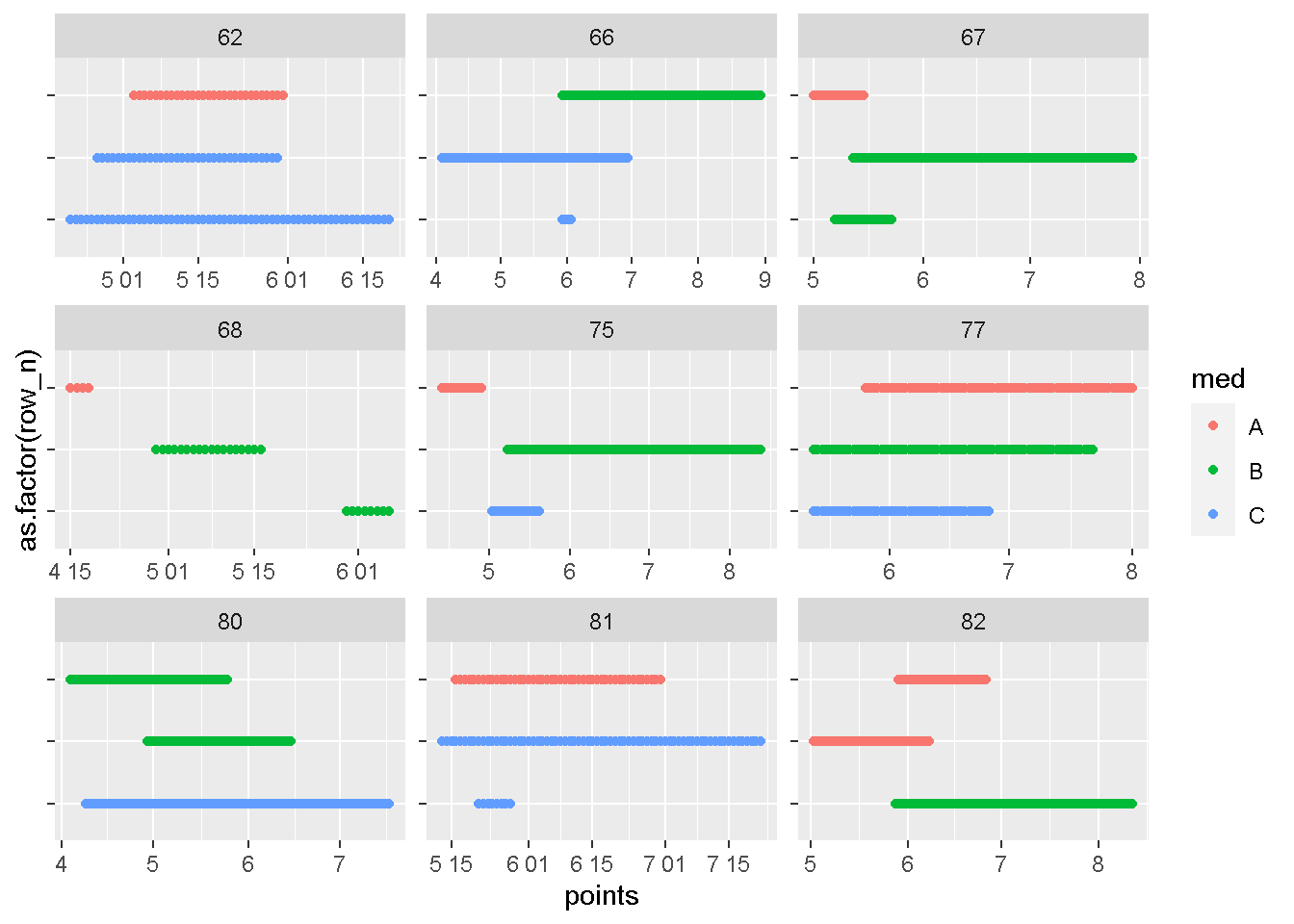
plot_id(dat,id_with_gt2r[28:36]) 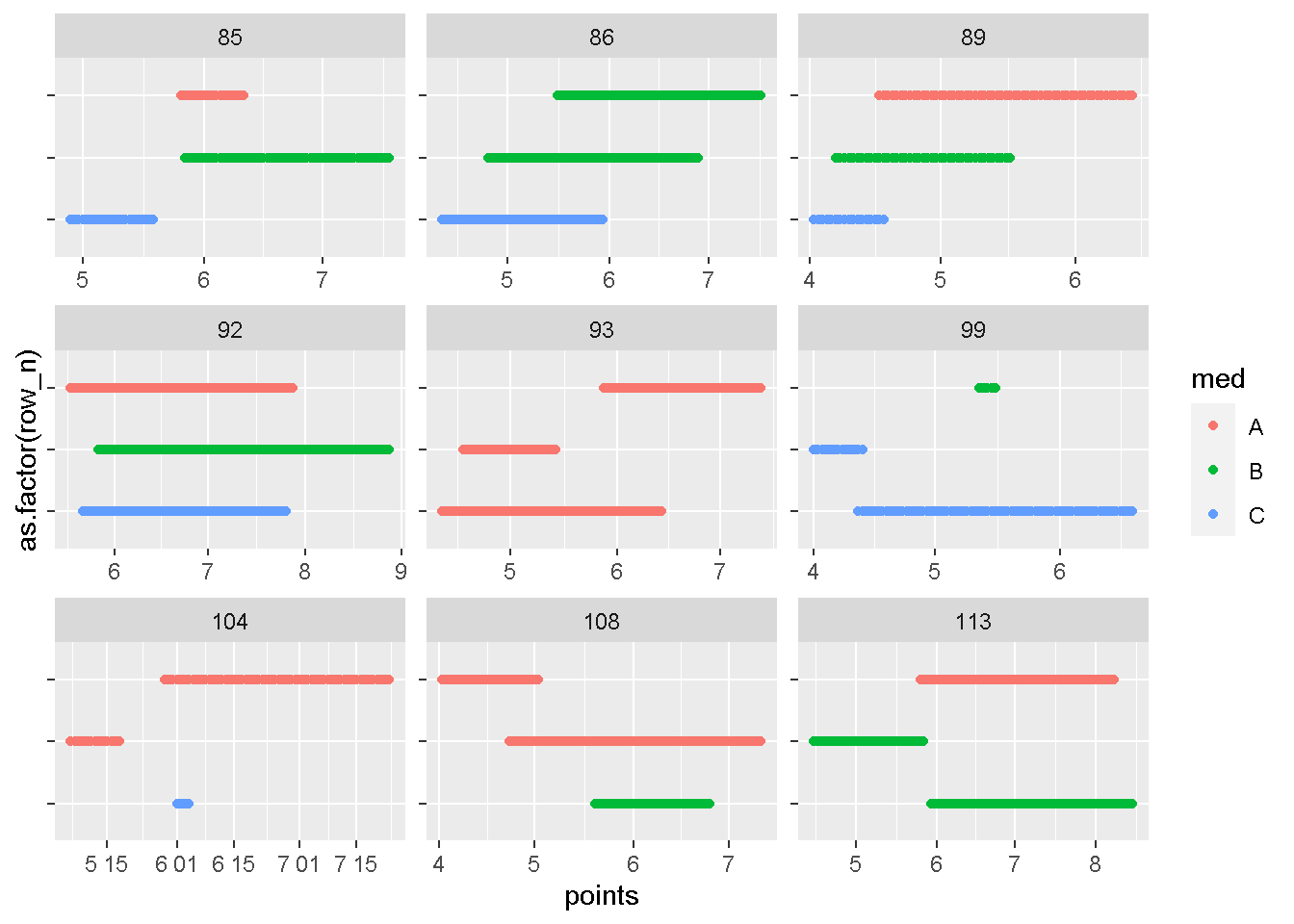
plot_id(dat,id_with_gt2r[37:45]) 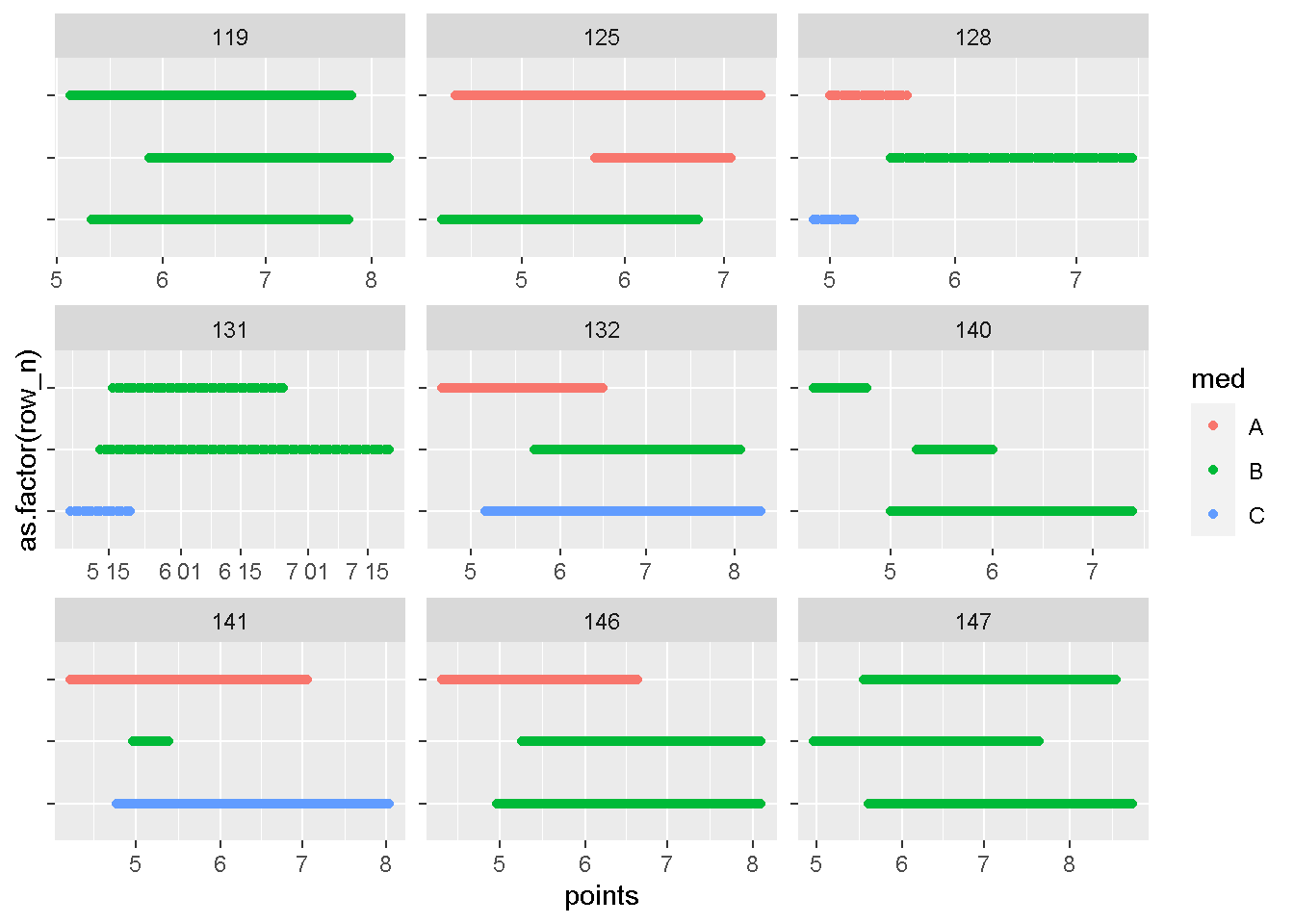
plot_id(dat,id_with_gt2r[46:54]) 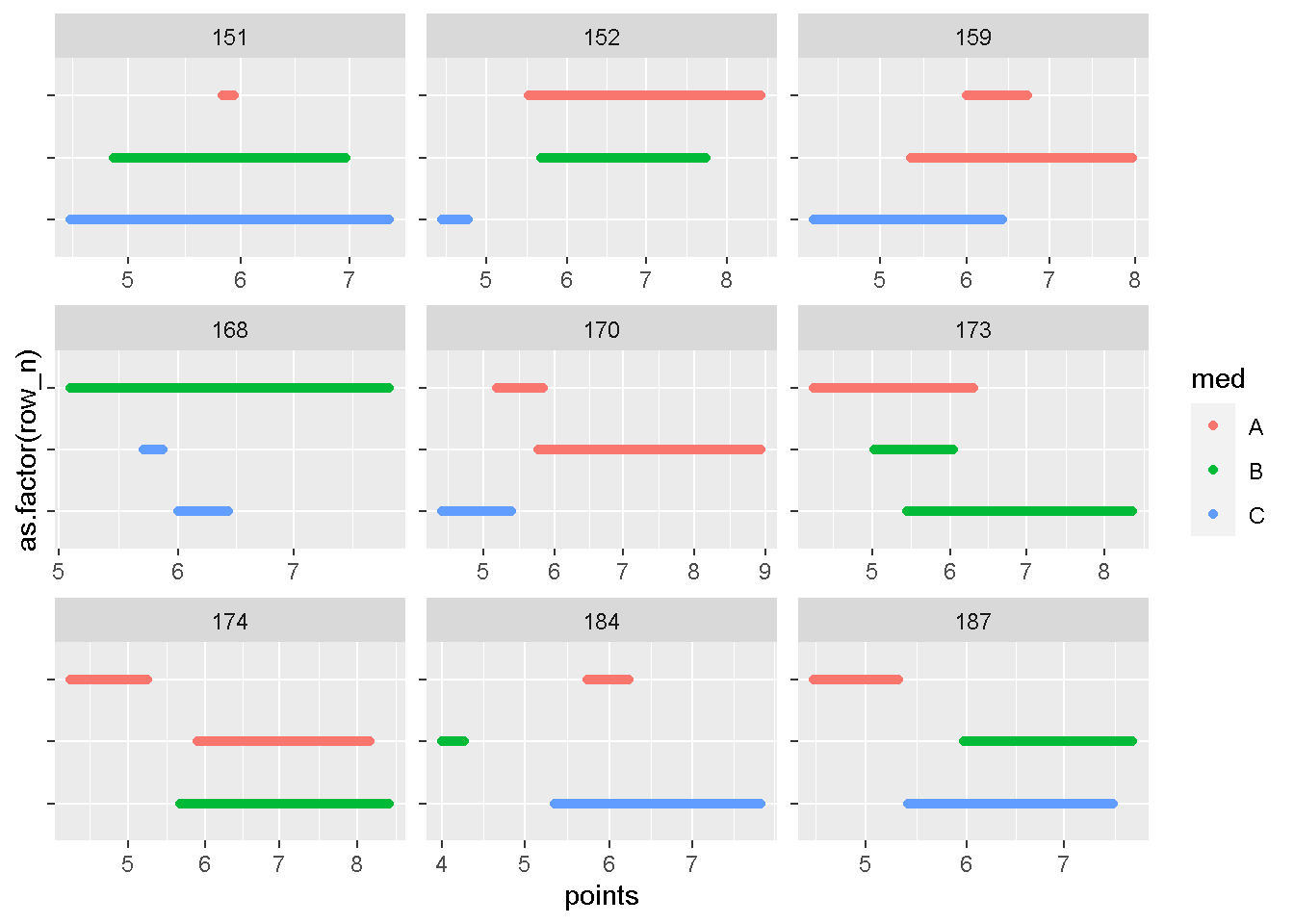
plot_id(dat,id_with_gt2r[55:61])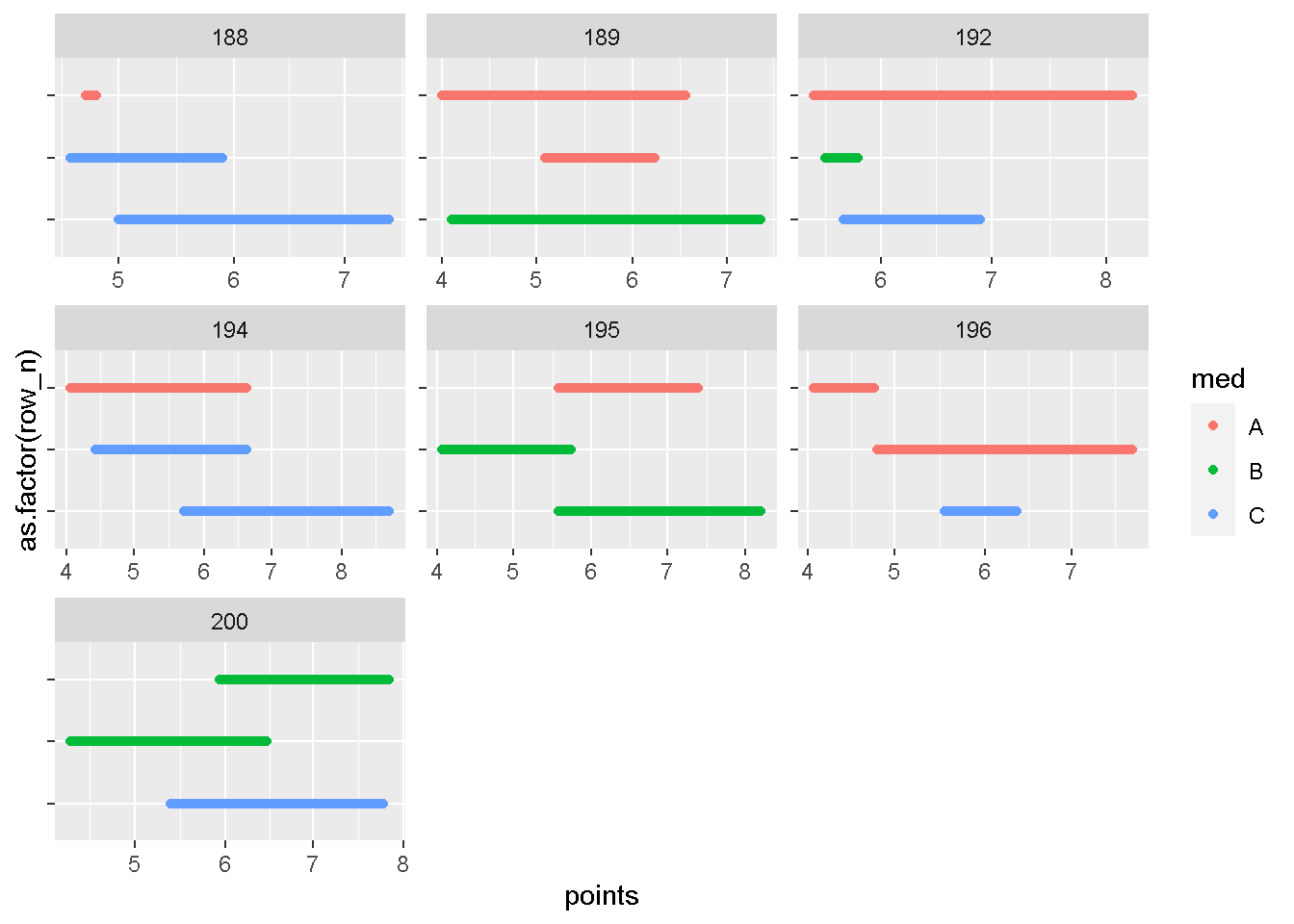
それでは、スライドでお示ししたスクリプトで処理を行っていきましょう
ここでは、
plot_id(dat,id_with_gt2r[11])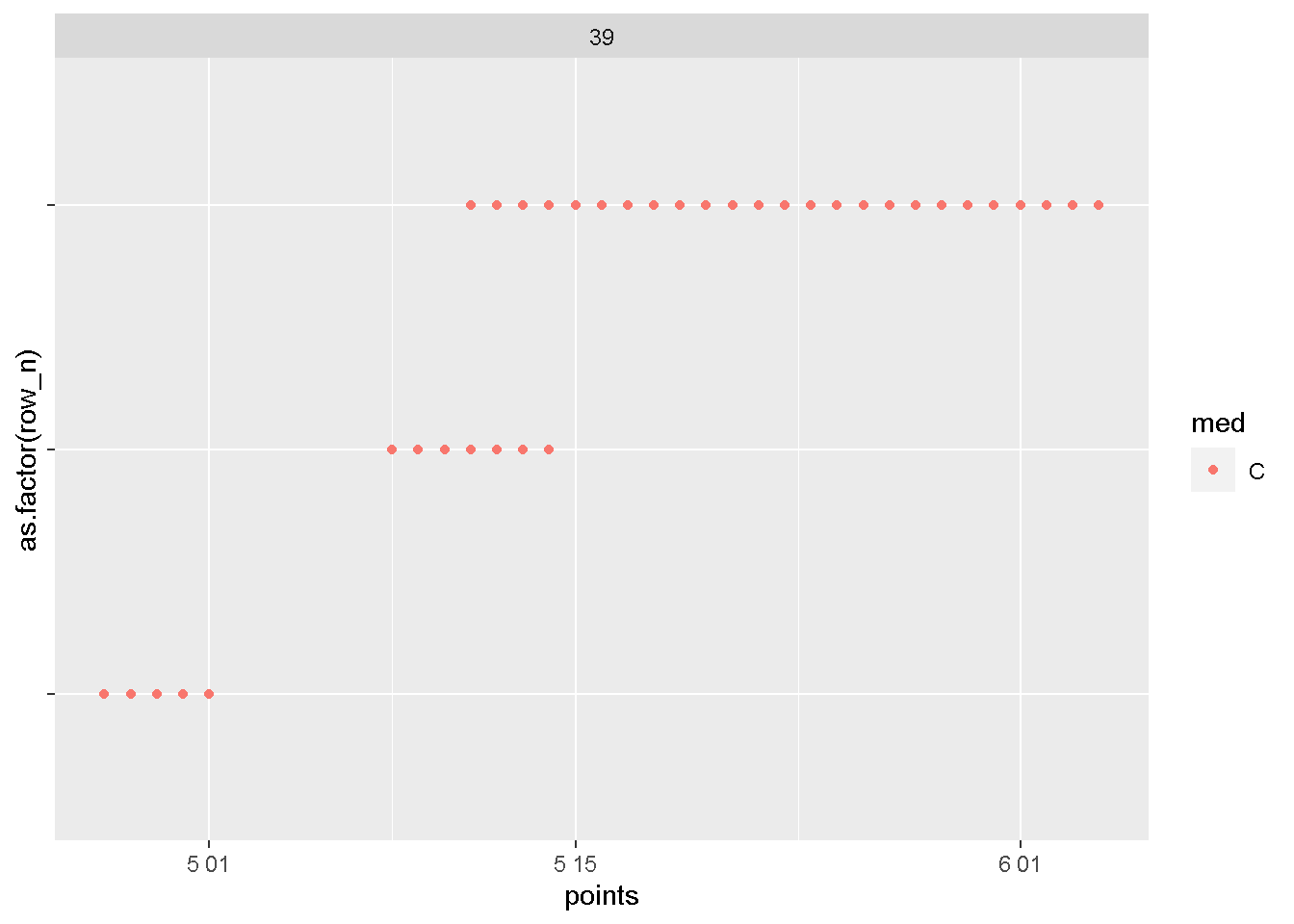
このデータをきれいにすることをまずは考えます。
temp <- dat %>%
filter(id == 39) %>%
select(start,end)ID39 は薬剤Cについて3つの期間が含まれるデータなので、スライドでの解説で行った形のデータになっています。
temp %>%
arrange(start) %>% #並びかえて
mutate(interv = interval(start, end)) %>% #intervalを作成して
mutate(
prev_overlap = int_overlaps(interv, lag(interv)),
prev_nextday = int_overlaps(interv, int_shift(lag(interv),days(1))),
) %>% #期間の重なりを確認
mutate(prev_oa = prev_overlap | prev_nextday) %>% #2条件を1つにまとめて
replace_na(list(prev_oa = FALSE)) %>% #欠損はFALSEで置き換え
mutate(presc_id = cumsum(!prev_oa)) %>% #TRUE FALSEをひっくり返して累積和
group_by(presc_id) %>% # 累積和(塊)毎にグループを作って
summarise(start = min(start), end = max(end)) # 各グループの最小・最大で集計する# A tibble: 2 × 3
presc_id start end
<int> <date> <date>
1 1 2020-04-27 2020-05-01
2 2 2020-05-08 2020-06-04と、こんな感じでうまく、5月1日までの塊と、5月8日移行の塊に分けることができました。
この処理、同じIDの同じ薬内での処理になっていますが、これを別々のIDと薬剤に処理してあげる必要があります。
plot_id(dat,id_with_gt2r[1:8])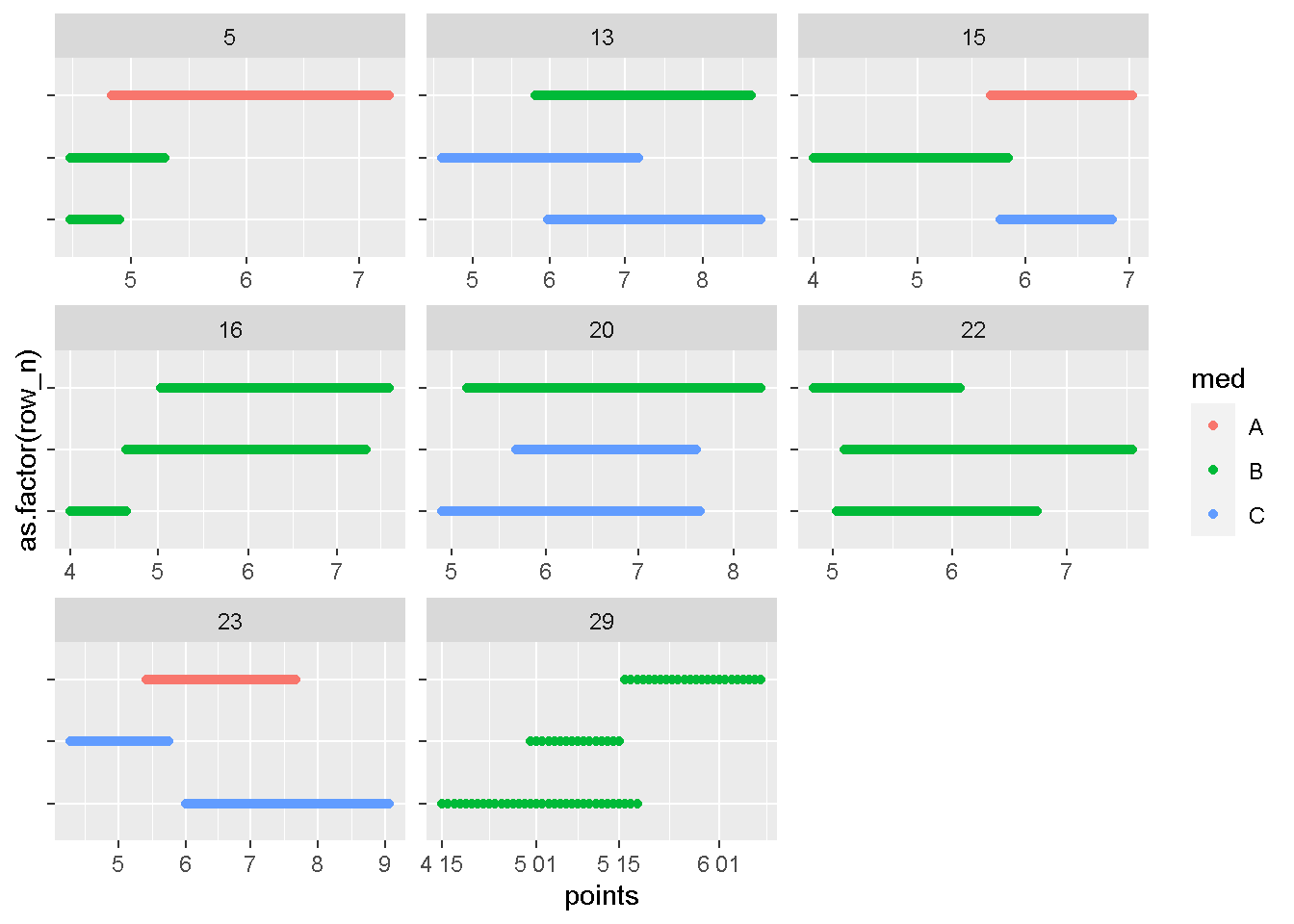
上で書いたスクリプトは、それぞれのID内で、同じ薬剤での処理となります。
ここで課題です。上のスクリプトを利用して、ID毎、ID毎の薬剤毎に上のスクリプトを適応する処理を書くことはできますか?
次の動画でIDと薬剤を加味したスクリプトを解説していきますが、できればまずご自身で可能か、取り組んでみてください。